Einführung
Das rotes neuronalesNeuronale Netze sind Rechenmodelle, die von der Funktionsweise des menschlichen Gehirns inspiriert sind. Sie nutzen Strukturen, die als künstliche Neuronen bekannt sind, um Daten zu verarbeiten und daraus zu lernen. Diese Netze sind grundlegend im Bereich der künstlichen Intelligenz, Dies ermöglicht erhebliche Fortschritte bei Aufgaben wie der Bilderkennung, Verarbeitung natürlicher Sprache und Vorhersage von Zeitreihen, unter anderen. Ihre Fähigkeit, komplexe Muster zu erlernen, macht sie zu mächtigen Werkzeugen.. Es handelt sich um eine Informationsverarbeitungsmaschine, die analog zum menschlichen Nervensystem betrachtet werden kann. Wie das menschliche Nervensystem, die aus miteinander verbundenen Neuronen besteht, Ein neuronales Netz besteht aus miteinander verbundenen Informationsverarbeitungseinheiten. Informationsverarbeitungseinheiten arbeiten nicht linear. Eigentlich, Das neuronale Netz erhält seine Stärke aus der parallelen Verarbeitung von Informationen, damit Sie mit Nichtlinearität umgehen können. Das neuronale Netz wird nützlich, um Bedeutungen abzuleiten und Muster aus komplexen Datensätzen zu erkennen.
Das neuronale Netz gilt als eine der nützlichsten Techniken in der Welt der AnalyseAnalytics bezieht sich auf den Prozess des Sammelns, Messen und analysieren Sie Daten, um wertvolle Erkenntnisse zu gewinnen, die die Entscheidungsfindung erleichtern. In verschiedenen Bereichen, wie Business, Gesundheit und Sport, Analysen können Muster und Trends erkennen, Prozesse optimieren und Ergebnisse verbessern. Der Einsatz fortschrittlicher Werkzeuge und statistischer Techniken ist unerlässlich, um Daten in anwendbares und strategisches Wissen umzuwandeln.... von Dateien. Aber trotzdem, ist komplex und wird oft als Blackbox angesehen, nämlich, Nutzer sehen die Ein- und Ausgabe eines neuronalen Netzes, haben aber keine Ahnung vom Prozess der Wissensgenerierung. Wir hoffen, dass der Artikel den Lesern hilft, den internen Mechanismus eines neuronalen Netzes kennenzulernen und praktische Erfahrungen zu sammeln, um ihn in R . zu implementieren.
Inhaltsverzeichnis
- Die Grundlagen des neuronalen Netzes
- Neuralnetzwerk-Tuning in R
- Kreuzvalidierung eines neuronalen Netzes
Die Grundlagen des neuronalen Netzes
Ein neuronales Netz ist ein Modell, das durch eine WeckfunktionDie Aktivierungsfunktion ist eine Schlüsselkomponente in neuronalen Netzen, da es den Output eines Neurons auf der Grundlage seiner Eingabe bestimmt. Sein Hauptzweck besteht darin, Nichtlinearitäten in das Modell einzuführen, Ermöglicht das Erlernen komplexer Muster in Daten. Es gibt verschiedene Aktivierungsfunktionen, wie das Sigmoid, ReLU und tanh, jedes mit besonderen Eigenschaften, die sich auf die Leistung des Modells in verschiedenen Anwendungen auswirken...., die von miteinander verbundenen Informationsverarbeitungseinheiten verwendet wird, um Eingaben in Ausgaben umzuwandeln. Ein neuronales Netz wurde schon immer mit dem menschlichen Nervensystem verglichen. Informationen passieren miteinander verbundene Einheiten in einer Weise, die analog zur Weitergabe von Informationen durch Neuronen beim Menschen ist.. Die erste Schicht des neuronalen Netzes erhält die Roheingaben, verarbeitet es und gibt die verarbeiteten Informationen an versteckte Schichten weiter. Die versteckte Schicht übergibt die Informationen an die letzte Schicht, was die Ausgabe erzeugt. Der Vorteil des neuronalen Netzes besteht darin, dass es von Natur aus adaptiv ist. Lernen Sie aus den bereitgestellten Informationen, nämlich, trainiert sich aus Daten, die ein bekanntes Ergebnis haben und ihre Gewichtung für eine bessere Vorhersage in Situationen mit unbekanntem Ergebnis optimiert.
Ein Perzeptron, nämlich. einschichtiges neuronales Netz, ist die einfachste Form eines neuronalen Netzes. Ein Perzeptron empfängt mehrdimensionale Informationen und verarbeitet sie durch eine gewichtete Summe und Aktivierungsfunktion. Es wird durch einen Lernalgorithmus und markierte Daten trainiert, die die Gewichte im Summationsprozessor optimieren. Eine wesentliche Einschränkung des Perzeptronmodells ist seine Unfähigkeit, mit Nichtlinearität umzugehen. Ein mehrschichtiges neuronales Netzwerk überwindet diese Einschränkung und hilft bei der Lösung nichtlinearer Probleme.. Das EingabeschichtDas "Eingabeschicht" bezieht sich auf die Anfangsebene in einem Datenanalyseprozess oder in neuronalen Netzwerkarchitekturen. Seine Hauptfunktion besteht darin, Rohinformationen zu empfangen und zu verarbeiten, bevor sie von nachfolgenden Schichten transformiert werden. Im Kontext des maschinellen Lernens, Die richtige Konfiguration der Eingabeschicht ist entscheidend, um die Effektivität des Modells zu gewährleisten und seine Leistung bei bestimmten Aufgaben zu optimieren.... Verbindet sich mit der verborgenen Schicht, die wiederum mit dem Ausgabe-LayerDas "Ausgabe-Layer" ist ein Konzept, das im Bereich der Informationstechnologie und des Systemdesigns verwendet wird. Es bezieht sich auf die letzte Schicht eines Softwaremodells oder einer Architektur, die für die Präsentation der Ergebnisse für den Endbenutzer verantwortlich ist. Diese Schicht ist entscheidend für die Benutzererfahrung, Da es eine direkte Interaktion mit dem System und die Visualisierung der verarbeiteten Daten ermöglicht..... Verbindungen werden gewichtet und Gewichtungen mit einer Lernregel optimiert.
Es gibt viele Lernregeln, die mit dem neuronalen Netz verwendet werden:
ein) kleinstes mittleres Quadrat;
B) Rückgang von SteigungGradient ist ein Begriff, der in verschiedenen Bereichen verwendet wird, wie Mathematik und Informatik, um eine kontinuierliche Variation von Werten zu beschreiben. In Mathematik, bezieht sich auf die Änderungsrate einer Funktion, während des Studiums im Grafikdesign, Gilt für den Farbübergang. Dieses Konzept ist unerlässlich, um Phänomene wie die Optimierung von Algorithmen und die visuelle Darstellung von Daten zu verstehen, ermöglicht eine bessere Interpretation und Analyse in...;
C) Newtonsche Regel;
D) Farbverlauf konjugieren, etc.
Die Lernregeln können in Verbindung mit der Methode des umgekehrten Ausbreitungsfehlers verwendet werden. Mit der Lernregel wird der Fehler in der Ausgabeeinheit berechnet. Dieser Fehler breitet sich rückwärts auf alle Antriebe aus, so dass der Fehler in jedem Antrieb proportional zum Beitrag dieses Antriebs zum Gesamtfehler im Ausgangsantrieb ist.. Fehler in jeder Einheit werden verwendet, um das Gewicht an jeder Verbindung zu optimieren. Das Abbildung"Abbildung" ist ein Begriff, der in verschiedenen Zusammenhängen verwendet wird, Von der Kunst zur Anatomie. Im künstlerischen Bereich, bezieht sich auf die Darstellung menschlicher oder tierischer Formen in Skulpturen und Gemälden. In der Anatomie, bezeichnet die Form und Struktur des Körpers. Was ist mehr, in der Mathematik, "Abbildung" Es hängt mit geometrischen Formen zusammen. Seine Vielseitigkeit macht es zu einem grundlegenden Konzept in mehreren Disziplinen.... 1 zeigt die Struktur eines einfachen neuronalen Netzmodells zum besseren Verständnis.
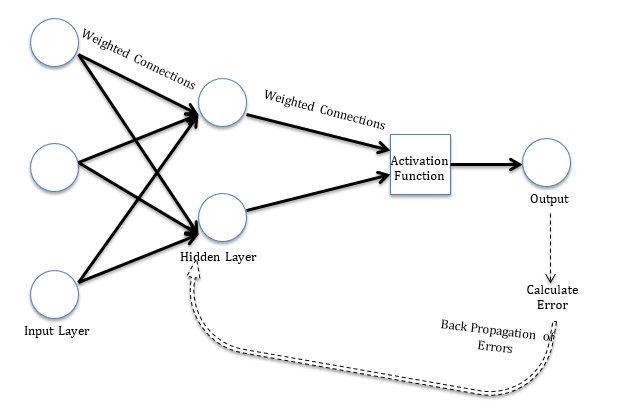
Abbildung 1 Ein einfaches neuronales Netzmodell
Neuralnetzwerk-Tuning in R
Nun passen wir ein neuronales Netzmodell in R . an. In diesem Artikel, wir verwenden eine Teilmenge des Getreidedatensatzes, der von der Carnegie Mellon University geteilt wird (CMU). Details zum Datensatz finden Sie unter folgendem Link: http://lib.stat.cmu.edu/DASL/Datafiles/Cereals.html. Ziel ist es, die Klassifizierung von Getreidevariablen als Kalorien vorherzusagen, Protein, Fette, etc. Das R-Skript wird nebeneinander bereitgestellt und zum besseren Verständnis des Benutzers kommentiert. . Die Daten liegen im .csv-Format vor und können durch Anklicken heruntergeladen werden: Getreide.
Legen Sie das Arbeitsverzeichnis in R mit . fest einstellen () Funktion und halten Sie Getreide.csv im Arbeitsverzeichnis. Wir verwenden das Rating als VariableIn Statistik und Mathematik, ein "Variable" ist ein Symbol, das einen Wert darstellt, der sich ändern oder variieren kann. Es gibt verschiedene Arten von Variablen, und qualitativ, die nicht-numerische Eigenschaften beschreiben, und quantitative, numerische Größen darstellen. Variablen sind grundlegend in Experimenten und Studien, da sie die Analyse von Beziehungen und Mustern zwischen verschiedenen Elementen ermöglichen, das Verständnis komplexer Phänomene zu erleichtern.... abhängig und Kalorien, Protein, Fette, Natrium und Ballaststoffe als unabhängige Variablen. Wir unterteilen die Daten in AusbildungTraining ist ein systematischer Prozess zur Verbesserung der Fähigkeiten, körperliche Kenntnisse oder Fähigkeiten. Es wird in verschiedenen Bereichen angewendet, wie Sport, Aus- und Weiterbildung. Zu einem effektiven Trainingsprogramm gehört auch die Zielplanung, Regelmäßiges Üben und Bewerten der Fortschritte. Anpassung an individuelle Bedürfnisse und Motivation sind Schlüsselfaktoren, um in jeder Disziplin erfolgreiche und nachhaltige Ergebnisse zu erzielen.... und Testset. Der Trainingssatz wird verwendet, um die Beziehung zwischen den abhängigen und unabhängigen Variablen zu finden, während der Testsatz die Leistung des Modells bewertet. Wir nehmen das 60% des Datensatzes als Trainingssatz. Die Zuordnung der Daten zum Trainings- und Testset erfolgt stichprobenartig. Wir führen eine Zufallsstichprobe in R durch mit Stichprobe ( ) Funktion. Wir haben benutzt set.seed () jedes Mal dieselbe Stichprobe zu generieren und Konsistenz bewahren. Wir werden die verwenden IndexDas "Index" Es ist ein grundlegendes Werkzeug in Büchern und Dokumenten, Dies ermöglicht es Ihnen, die gewünschten Informationen schnell zu finden. Allgemein, Sie wird am Anfang einer Arbeit präsentiert und organisiert die Inhalte hierarchisch, mit Kapiteln und Abschnitten. Die richtige Vorbereitung erleichtert die Navigation und verbessert das Verständnis des Materials, was es zu einer unverzichtbaren Ressource sowohl für Studenten als auch für Fachleute in verschiedenen Bereichen macht.... variabel, während das neuronale Netz optimiert wird, um Test- und Trainingsdatensätze zu erstellen. Das R-Skript lautet wie folgt:
## Indexvariable erstellen
# Lesen Sie die Daten
data = read.csv("Getreide.csv", Kopfzeile=T)
# Stichproben
Stichprobenumfang = 0.60 * jetzt(Daten)
set.seed(80)
Index = Probe( seq_len ( jetzt ( Daten ) ), Größe = Mustergröße )
# Trainings- und Testset erstellen
Datenzug = Daten[ Index, ]
Datentest = Daten[ -Index, ]
Jetzt passen wir ein neuronales Netz an unsere Daten an. Wir gebrauchen neuronales Netz Bibliothek zur Analyse. Der erste Schritt besteht darin, den Getreidedatensatz zu skalieren. Der Umfang der Daten ist wichtig, weil, andererseits, eine Variable kann allein aufgrund ihrer Skalierung einen großen Einfluss auf die Prädiktorvariable haben. Die Verwendung ohne Skala kann zu bedeutungslosen Ergebnissen führen. Gängige Techniken zum Skalieren von Daten sind: StandardisierungNormung ist ein grundlegender Prozess in verschiedenen Disziplinen, , die darauf abzielt, einheitliche Standards und Kriterien zur Verbesserung von Qualität und Effizienz festzulegen. In Kontexten wie dem Ingenieurwesen, Bildung und Verwaltung, Standardisierung erleichtert den Vergleich, Interoperabilität und gegenseitiges Verständnis. Bei der Implementierung von Standards, Der Zusammenhalt wird gefördert und die Ressourcen werden optimiert, die zu einer nachhaltigen Entwicklung und zur kontinuierlichen Verbesserung der Prozesse beiträgt.... Minimum-Maximum, Z-Score-Normalisierung, MedianDer Median ist ein statistisches Maß, das den zentralen Wert eines Satzes geordneter Daten darstellt. Um es zu berechnen, Die Daten werden von der niedrigsten zur höchsten sortiert und die Zahl in der Mitte wird identifiziert. Wenn es eine gerade Anzahl von Beobachtungen gibt, Die beiden Kernwerte werden gemittelt. Dieser Indikator ist besonders nützlich bei asymmetrischen Verteilungen, da es nicht von Extremwerten beeinflusst wird.... und MAD- und tan-h-Schätzer. Die Minimum-Maximum-Normalisierung transformiert die Daten in einen gemeinsamen Bereich, wodurch der Skaleneffekt aller Variablen eliminiert wird. Im Gegensatz zur Normalisierung des Z-Scores und der Methode des Medians und MAD, die Minimum-Maximum-Methode bewahrt die ursprüngliche Verteilung der Variablen. Wir verwenden Minimum-Maximum-Normalisierung, um die Daten zu skalieren. Das R-Skript zum Skalieren der Daten lautet wie folgt.
## Daten für neuronale Netze skalieren max = anwenden(Daten , 2 , max) min = anwenden(Daten, 2 , Mindest) skaliert = as.data.frame(Skala(Daten, Mitte = min, Skala = max - Mindest))
Skalierte Daten werden verwendet, um sich an das neuronale Netz anzupassen. Wir visualisieren das neuronale Netz mit Gewichten für jede der Variablen. Das R-Skript lautet wie folgt.
## Neuronales Netz anpassen
# Bibliothek installieren
install.pakete("neuronales Netz ")
# Bibliothek laden
Bücherei(neuronales Netz)
# Trainings- und Testset erstellen
trainNN = skaliert[Index , ]
testNN = skaliert[-Index , ]
# Fit neuronales Netz
set.seed(2)
NN = neuronales Netz(Bewertung ~ Kalorien + Protein + Fett + Natrium + Faser, ZugNN, versteckt = 3 , linearer.Ausgang = T )
# neuronales Netz zeichnen
Handlung(NN)
Die Figur 3 visualisieren Sie das berechnete neuronale Netz. Unser Modell hat 3 Neuronen in seiner verborgenen Schicht. Die schwarzen Linien zeigen die Verbindungen mit Gewichten. Gewichte werden mit dem oben erläuterten Backpropagation-Algorithmus berechnet. Die blaue Linie zeigt den Bias-Term.
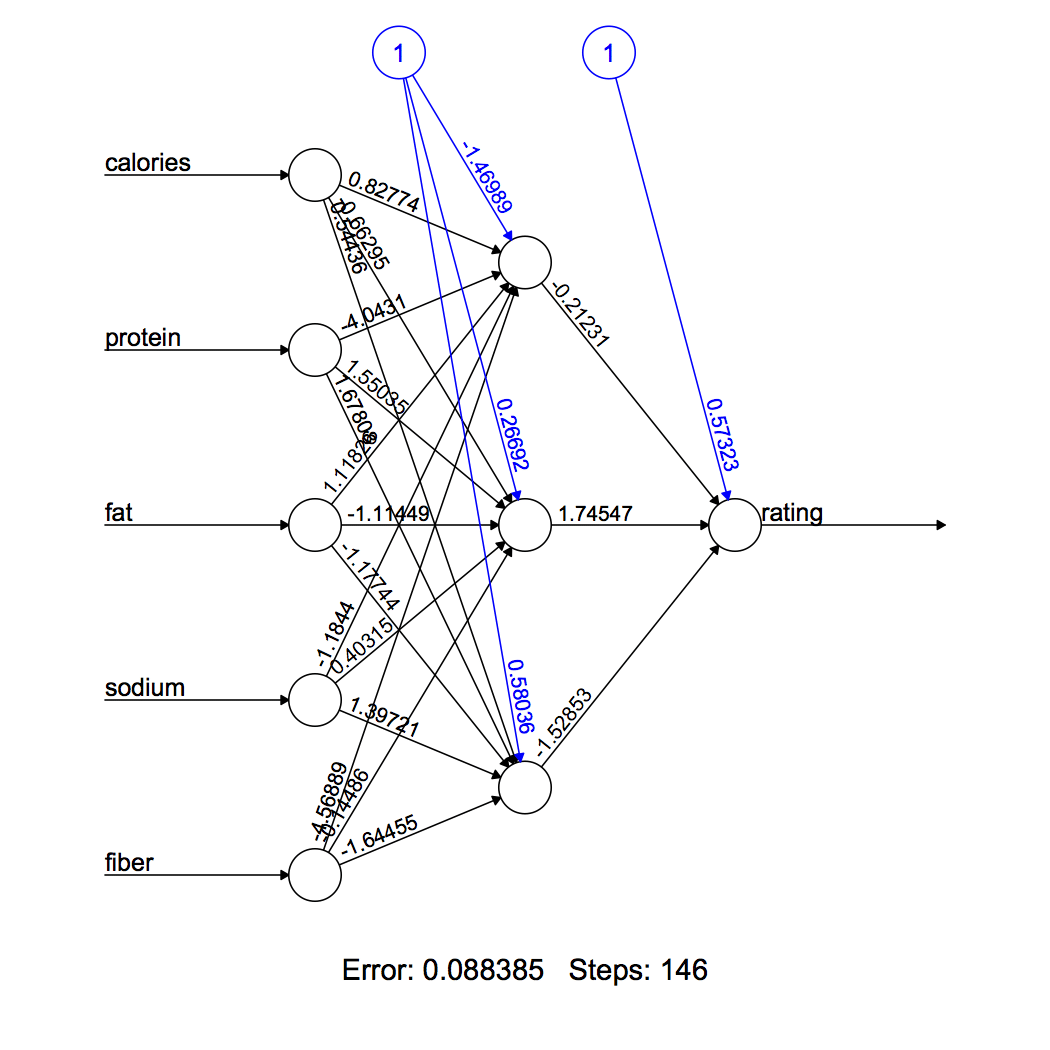
Abbildung 2 Rotes Neuron
Wir sagen die Bewertung mit dem neuronalen Netzmodell voraus. Der Leser muss sich daran erinnern, dass die prognostizierte Bewertung skaliert wird und muss sie transformieren, um einen Vergleich mit der tatsächlichen Bewertung anstellen zu können. Wir vergleichen auch die prognostizierte Bewertung mit der tatsächlichen Bewertung, indem wir visualisieren. Der RMSE für das neuronale Netzmodell ist 6.05. Der Leser kann in einem anderen Artikel mehr über RMSE erfahren, die durch Anklicken erreichbar sind hier. Das R-Skript lautet wie folgt:
## Vorhersage mit neuronalen Netzen vorhersagen_testNN = berechnen(NN, testNN[,C(1:5)]) Predict_testNN = (Predict_testNN$net.result * (max(data$rating) - Mindest(data$rating))) + Mindest(data$rating) Handlung(Datentest $ Bewertung, Predict_testNN, col="Blau", pc=16, ylab = "prognostizierte Bewertung NN", xlab = "echte Bewertung") abline(0,1) # Berechnen des mittleren quadratischen Fehlers (RMSE) RMSE.NN = (Summe((Datentest $ Bewertung - Predict_testNN)^2) / jetzt(Datentest)) ^ 0.5
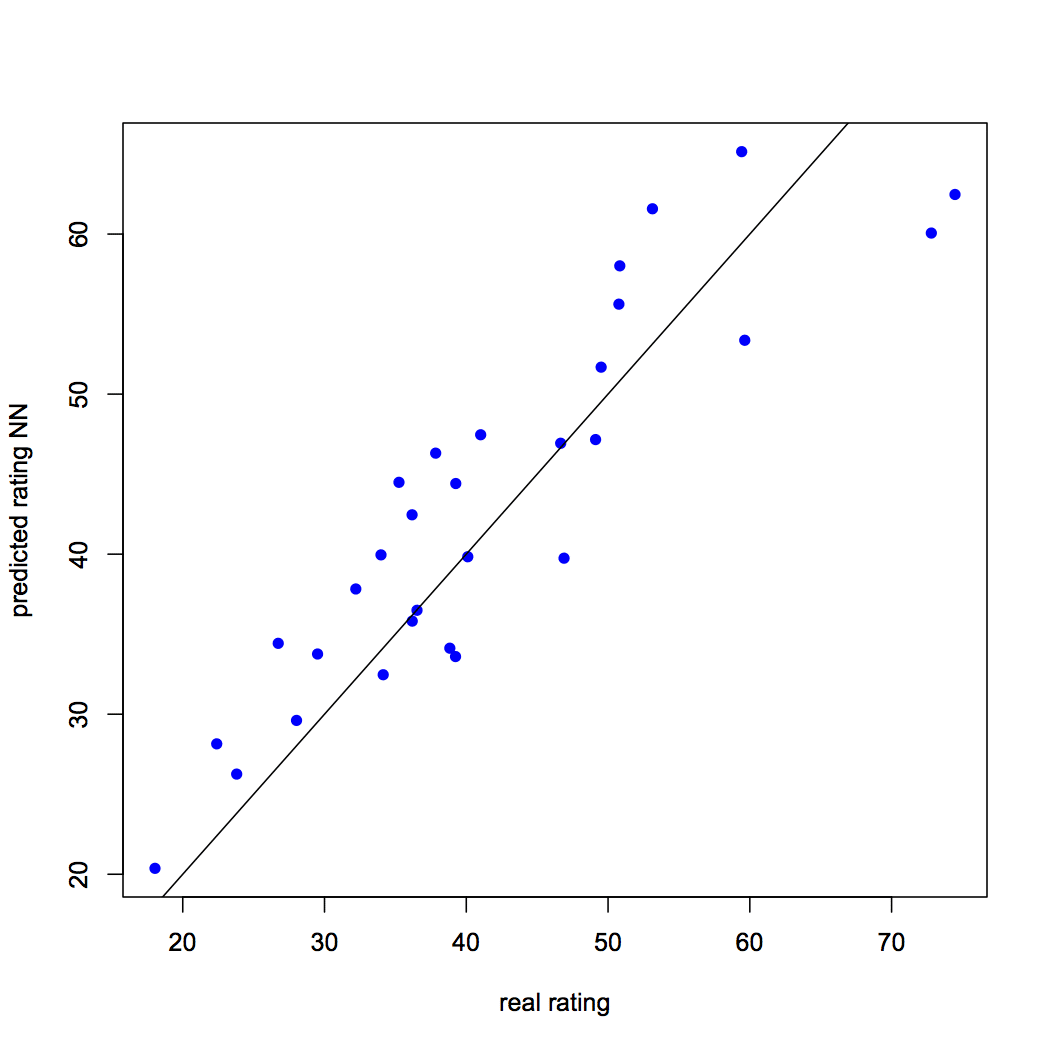
Abbildung 3: Vorhergesagte vs. tatsächliche Bewertung mithilfe eines neuronalen Netzes
Kreuzvalidierung eines neuronalen Netzes
Wir haben unsere neuronale Netzmethode mit RMSE evaluiert, Was ist eine Residualbewertungsmethode?. Das Hauptproblem bei Residualbewertungsmethoden besteht darin, dass sie uns nicht über das Verhalten unseres Modells bei der Einführung neuer Daten informieren.. Wir versuchen, das Problem von . zu lösen „neue Daten“ Aufteilung unserer Daten in Trainings- und Test-Set, Aufbau des Modells im Trainingssatz und Bewertung des Modells Berechnung des RMSE für den Testsatz. Der Trainingstest-Split war nichts anderes als die einfachste Form der Kreuzvalidierungsmethode, bekannt als Aufbewahrungsmethode. Eine Einschränkung der Aufbewahrungsmethode ist die Varianz der Leistungsbewertungsmetrik, in unserem Fall RMSE, kann hoch sein, basierend auf den Elementen, die dem Trainings- und Testset zugewiesen sind.
Die zweite Kreuzvalidierungstechnik ist üblicherweise Kreuzvalidierung von k-fold. Diese Methode kann als wiederkehrend angesehen werden Aufbewahrungsmethode. Die vollständigen Daten werden in k gleiche Teilmengen unterteilt und jedes Mal, wenn eine Teilmenge als Testmenge zugewiesen wird, andere werden verwendet, um das Modell zu trainieren. Jeder Datenpunkt hat die Chance, im Test-Set und im Trainings-Set zu sein, Diese Methode reduziert also die Abhängigkeit der Leistung von der Test-Trainingsabteilung und reduziert die Varianz der Leistungsmetriken. Der Extremfall von Kreuzvalidierung von k-fold tritt auf, wenn k gleich der Anzahl der Datenpunkte ist. Das würde bedeuten, dass das Vorhersagemodell an allen Datenpunkten außer einem Datenpunkt trainiert wird, die die Rolle eines Test-Sets übernimmt. Diese Methode, einen Datenpunkt als Testmenge zu belassen, ist bekannt als Hinterlasse eine Kreuzvalidierung.
Jetzt treten wir auf Kreuzvalidierung von k-fold in dem neuronalen Netzmodell, das wir im vorherigen Abschnitt erstellt haben. Die Anzahl der Elemente im Trainingsset, J, variieren von 10 ein 65 und für jeden J, Extrahiert 100 Datensatzproben. Die restlichen Elemente werden jeweils dem Testset zugeordnet. Das Modell wird in jedem der 5600 Trainingsdatensätze und dann auf den entsprechenden Testsätzen getestet. Wir berechnen den RMSE jedes der Testsätze. Die RMSE-Werte für jeden der Sätze werden in einem Array gespeichert.[100 x 56]. Diese Methode stellt sicher, dass unsere Ergebnisse frei von Stichprobenverzerrungen sind und verifiziert die Robustheit unseres Modells.. Wir verwenden verschachtelte for-Schleife. Das R-Skript lautet wie folgt:
## Kreuzvalidierung des neuronalen Netzmodells
# relevante Bibliotheken installieren
install.pakete("Stiefel")
install.pakete("Pflaume")
# Bibliotheken laden
Bücherei(Stiefel)
Bücherei(Pflaume)
# Variablen initialisieren
set.seed(50)
k = 100
RMSE.NN = NULL
Liste = Liste( )
# Anpassung des neuronalen Netzmodells an eine verschachtelte for-Schleife
zum(j in 10:65){
zum (ich bin dabei 1:k) {
Index = Probe(1:jetzt(Daten),J )
trainNN = skaliert[Index,]
testNN = skaliert[-Index,]
Datentest = Daten[-Index,]
NN = neuronales Netz(Bewertung ~ Kalorien + Protein + Fett + Natrium + Faser, ZugNN, versteckt = 3, lineare.Ausgabe= T)
vorhersagen_testNN = berechnen(NN,testNN[,C(1:5)])
Predict_testNN = (Predict_testNN$net.result*(max(data$rating)-Mindest(data$rating)))+Mindest(data$rating)
RMSE.NN [ich]<- (Summe((Datentest $ Bewertung - Predict_testNN)^2)/jetzt(Datentest))^0,5
}
Aufführen[[J]] = RMSE.NN
}
Matrix.RMSE = do.call(cbind, Aufführen)
Auf RMSE-Werte kann über die Variable Matrix.RMSE zugegriffen werden. Die Größe der Matrix ist groß; Daher, Wir werden versuchen, den Daten durch Visualisierungen einen Sinn zu geben. Zuerst, wir werden einen Boxplot für eine der Spalten in Matrix.RMSE vorbereiten, wobei der Trainingssatz eine Länge gleich hat 65. Diese können vorbereitet werden BoxplotsBox-Diagramme, Auch bekannt als Box- und Whisker-Diagramme, sind statistische Werkzeuge, die die Verteilung eines Datensatzes darstellen. Diese Diagramme zeigen den Median, Quartile und Ausreißer, Ermöglicht die Visualisierung von Datenvariabilität und -symmetrie. Sie sind nützlich für den Vergleich zwischen verschiedenen Gruppen und in der explorativen Analyse, So lassen sich Trends und Muster in den Daten leichter erkennen.... für jede der Trainingssatzlängen (10 ein 65). Das R-Skript lautet wie folgt.
## Boxplot vorbereiten Box-Plot(Matrix.RMSE[,56], ylab = "RMSE", Haupt = "RMSE BoxPlot (Länge des Trainingssatzes = 65)")
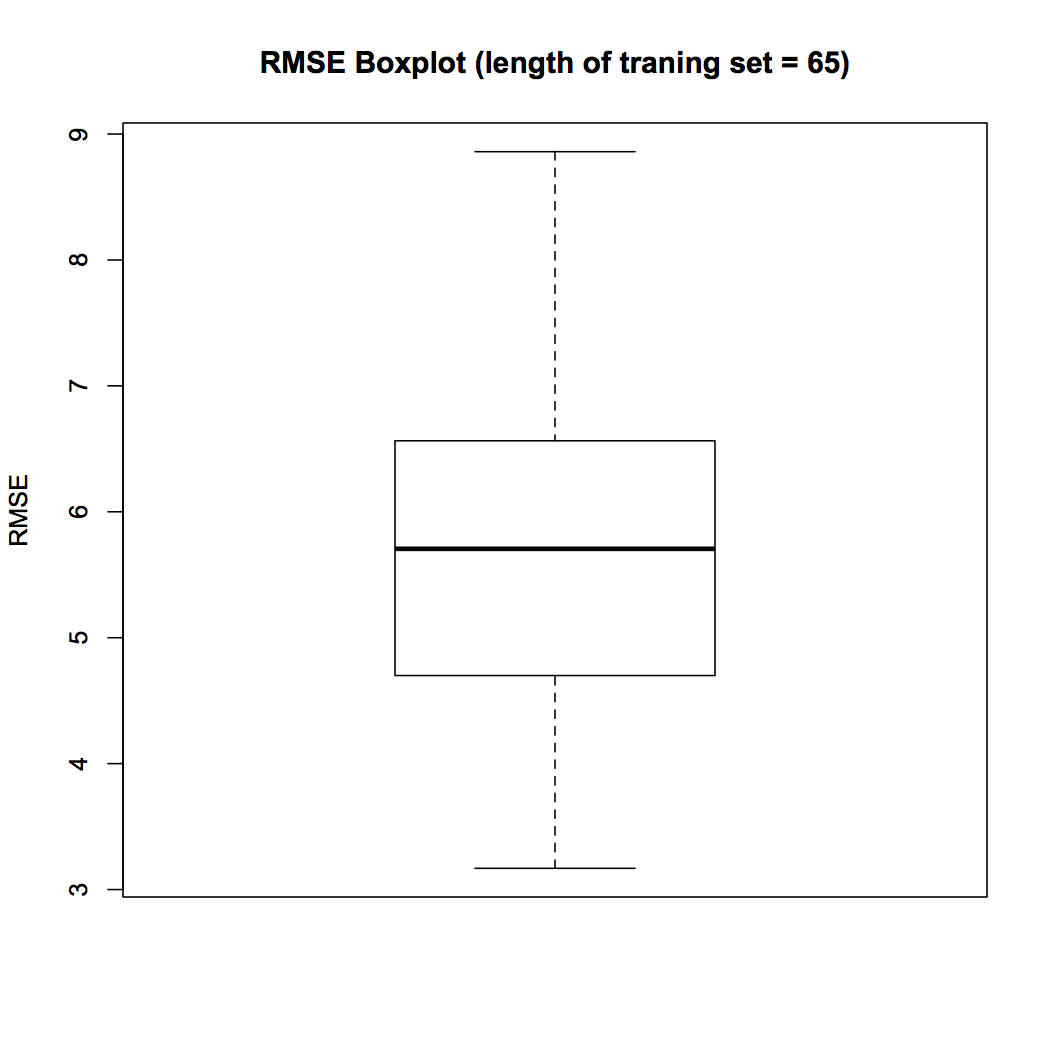
Abbildung 4 Box-Plot
Der Boxplot von Abb. 4 zeigt, dass der Median-RMSE in 100 Proben, wenn die Trainingssatzdauer auf eingestellt ist 65 es ist 5.70. In der folgenden Visualisierung, Wir untersuchen die Variation des RMSE mit der Dauer des Trainingssatzes. Wir berechnen den Median-RMSE für jede der Längen des Trainingssatzes und zeichnen sie mit dem folgenden R-Skript.
## Variation des Median-RMSE
install.pakete("MatrixStats")
Bücherei(MatrixStats)
med = colMedian(Matrix.RMSE)
X = seq(10,65)
Handlung (mit ~ X, Typ = "l", xlab = "Länge des Trainingssets", ylab = "Median-RMSE", Haupt = "Variation des RMSE mit der Länge des Trainingssatzes")
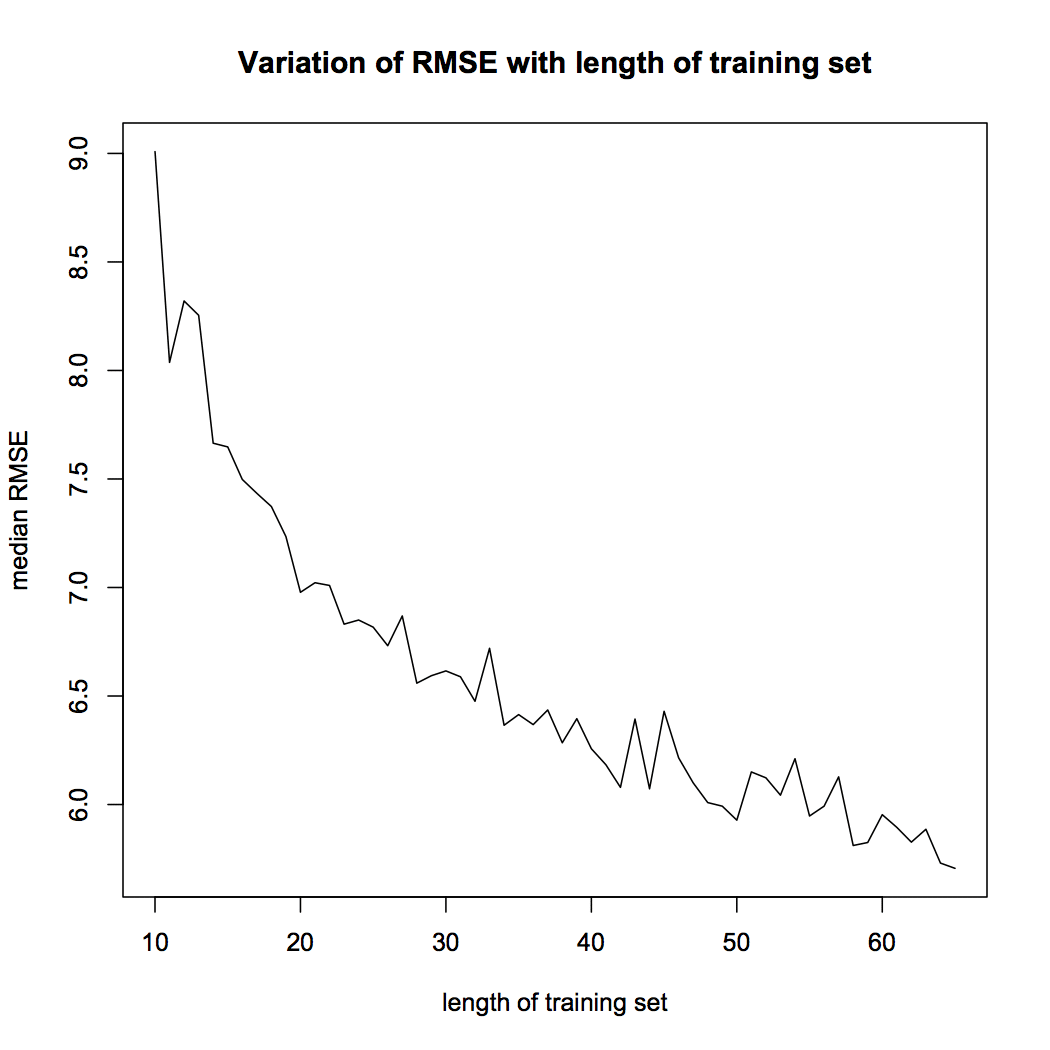
Abbildung 5 RMSE-Variation
Die Figur 5 zeigt, dass der mediane RMSE unseres Modells auf messenDas "messen" Es ist ein grundlegendes Konzept in verschiedenen Disziplinen, , die sich auf den Prozess der Quantifizierung von Eigenschaften oder Größen von Objekten bezieht, Phänomene oder Situationen. In Mathematik, Wird verwendet, um Längen zu bestimmen, Flächen und Volumina, In den Sozialwissenschaften kann es sich auf die Bewertung qualitativer und quantitativer Variablen beziehen. Die Messgenauigkeit ist entscheidend, um zuverlässige und valide Ergebnisse in der Forschung oder praktischen Anwendung zu erhalten.... als die Dauer der Serienschulung. Das ist ein wichtiges Ergebnis. Der Leser sollte daran denken, dass die Genauigkeit des Modells von der Dauer des Trainingssatzes abhängt.. Die Leistung des neuronalen Netzmodells hängt von der Trainingstestabteilung ab.
Abschließende Anmerkungen
Der Artikel analysiert die theoretischen Aspekte eines neuronalen Netzes, deren Umsetzung in R und Evaluation nach dem Training. Das neuronale Netz ist vom biologischen Nervensystem inspiriert. Ähnlich wie das Nervensystem, Informationen durchlaufen Schichten von Prozessoren. Die Bedeutung der Variablen wird durch die Gewichtung jeder Verbindung dargestellt. Der Artikel vermittelt ein grundlegendes Verständnis des Backpropagation-Algorithmus, die verwendet wird, um diese Gewichte zuzuweisen. In diesem Artikel implementieren wir auch ein neuronales Netz in R. Wir verwenden einen öffentlich zugänglichen Datensatz, der von CMU . geteilt wird. Ziel ist es, die Klassifizierung von Getreide anhand von Informationen wie Kalorien vorherzusagen, Fette, Protein, etc. Nach dem Aufbau des neuronalen Netzes, Wir bewerten die Präzision und Robustheit des Modells. Wir berechnen RMSE und führen Kreuzvalidierungsanalysen durch. In Kreuzvalidierung, Wir überprüfen die Variation in der Präzision des Modells, wenn sich die Dauer des Trainingssatzes ändert. Wir betrachten Trainingssätze mit einer Länge von 10 ein 65. Für jede Länge, werden zufällig ausgewählt 100 Stichproben und der Median-RMSE wird berechnet. Wir zeigen, dass die Genauigkeit des Modells zunimmt, wenn die Trainingsmenge groß ist. Bevor Sie das Modell für die Vorhersage verwenden, Es ist wichtig, die Robustheit der Leistung durch Kreuzvalidierung zu überprüfen.
Der Artikel bietet einen kurzen Überblick über das neuronale Netz und ist eine nützliche Referenz für Datenenthusiasten.. Wir haben im gesamten Artikel einen kommentierten R-Code bereitgestellt, um Lesern mit praktischer Erfahrung mit neuronalen Netzen zu helfen..
Bio: Chaitanya Sagar ist Gründer und CEO von Wahrnehmungsanalytik. Perceptive Analytics ist eines der führenden Analyseunternehmen in Indien. Arbeit in Marketing Analytics für E-Commerce-Unternehmen, Einzelhandel und Pharma.






