Cet article a été publié dans le cadre du Blogathon sur la science des données
L'ablation du crâne est l'une des étapes préliminaires à la détection d'anomalies dans le cerveau.. C'est le processus d'isolement du tissu cérébral du tissu non cérébral à partir d'une image IRM d'un cerveau. Cette segmentation du cerveau à partir du crâne est une tâche fastidieuse même pour les radiologues experts et la précision des résultats varie considérablement d'une personne à l'autre.. Ici, nous essayons d'automatiser le processus en créant un pipeline de bout en bout où nous avons seulement besoin d'entrer l'image IRM brute et le pipeline devrait générer une image segmentée du cerveau après avoir effectué le pré-traitement nécessaire..
Ensuite, Qu'est-ce qu'une image IRM?
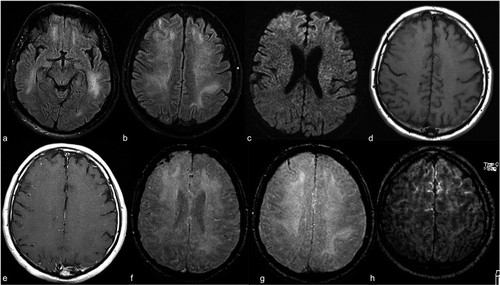
Pour obtenir une image IRM d'un patient, sont insérés dans un tunnel avec un champ magnétique à l'intérieur. Cela fait que tous les protons du corps “s'aligner”, donc son spin quantique est le même. Ensuite, une impulsion de champ magnétique oscillant est utilisée pour perturber cet alignement. Quand les protons reviennent à l'équilibre, envoyer une onde électromagnétique. Selon la teneur en matières grasses, la composition chimique et, ce qui est plus important, le type de stimulation (c'est-à-dire, les séquences) utilisé pour perturber les protons, différentes images seront obtenues. Quatre séquences communes qui sont obtenues sont T1, T1 avec contraste (T1C), T2, Oui INSTINCT.
Défis courants lorsque l'on travaille avec l'imagerie cérébrale
-
Les défis des données du monde réel
Construire un modèle et obtenir une bonne précision sur un ordinateur portable jupyter, c'est bien. Mais la plupart du temps, un modèle très performant fonctionne très mal sur les données du monde réel. Cela se produit en raison de la dérive des données lorsque le modèle voit des données complètement différentes de celles pour lesquelles il est formé.. Dans notre cas, cela peut arriver en raison de différences dans certains paramètres ou méthodes d'imagerie par résonance magnétique. Voici un Blog décrivant certains échecs de l'IA dans le monde réel.
Formulation du problème
La tâche que nous avons ici est de donner une image IRM 3D que nous devons identifier le cerveau et segmenter le tissu cérébral à partir de l'image complète d'un crâne.. Pour cette tâche, nous aurons une étiquette de vérité de base et, donc, sera une tâche de segmentation d'image supervisée. Nous utiliserons perte de dés comme notre fonction de perte.
Données
Jetons un coup d'œil à l'ensemble de données que nous utiliserons pour cette tâche. L'ensemble de données peut être téléchargé à partir de ici.
Le référentiel contient des données de 125 participants, de 21 une 45 ans, avec une variété de symptômes psychiatriques cliniques et subcliniques. Pour chaque participant, le référentiel contient:
- Imagerie par résonance magnétique structurelle pondérée en T1 (sans visage): il s'agit d'une image IRM brute pondérée en T1 avec un seul canal.
- Masque cérébral: est le masque d'image du cerveau ou peut-on l'appeler la vérité fondamentale. Obtenu en utilisant la méthode Beast (extraction cérébrale basée sur une segmentation non locale) et appliquer des modifications manuelles par des experts du domaine pour éliminer les tissus non cérébraux.
- Image crâne dépouillé: cela peut être considéré comme une partie du cerveau dépouillé de l'image précédente pondérée en T1. Ceci est similaire à la superposition de masques sur des images réelles.
La résolution des images est 1 mm3 et chaque fichier est au format NiFTI (.nii.gz). Un seul point de données ressemble à ceci …
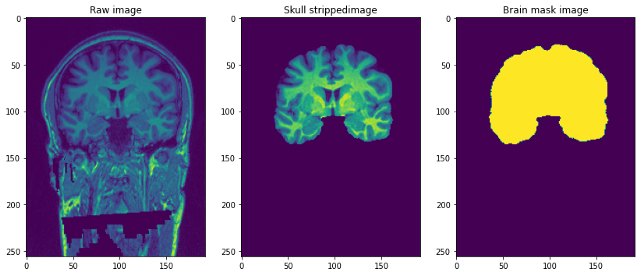
Prétraitement de nos images Raw
img=nib.load('/content/NFBS_Dataset/A00028185/sub-A00028185_ses-NFB3_T1w.nii.gz')
imprimer('Forme de l'image=",img.forme)

Imaginez des images 3D ci-dessus comme si nous avions 192 Images de taille 2D 256 * 256 empilés les uns sur les autres.
Créons un bloc de données qui contient l'emplacement des images et les masques correspondants et les images sans crânes.
#mémoriser l'adresse de 3 types de fichiers
importer le système d'exploitation
brain_mask=[]
cerveau =[]
brut =[]
pour sous-répertoire, dirs, fichiers dans os.walk("/contenu/NFBS_Dataset'):
pour le fichier dans les fichiers:
#imprimer os.path.join(sous-répertoire, déposer)oui
chemin de fichier = sous-répertoire + os.sep + déposer
si chemin_fichier.se termine par(".gz"):
si '_brainmask.' dans le chemin du fichier:
brain_mask.append(chemin du fichier)
elif '_brain.' dans le chemin du fichier:
cerveau.append(chemin du fichier)
autre:
raw.append(chemin du fichier)
Le signal du champ de polarisation est un signal basse fréquence très doux qui corrompt les images IRM., surtout celles produites par les anciennes IRM (Imagerie par résonance magnétique) Machines. Algorithmes de traitement d'images tels que la segmentation, l'analyse de la texture ou la classification à l'aide des valeurs de niveau de gris des pixels de l'image ne produira pas de résultats satisfaisants. Une étape de prétraitement est nécessaire pour corriger le signal de champ polarisant avant d'envoyer des images IRM corrompues à de tels algorithmes ou les algorithmes doivent être modifiés.
-
Recadrer et redimensionner
En raison des limitations de calcul de l'ajustement de l'image complète au modèle ici, Nous avons décidé de réduire la taille de l'image IRM de (256 * 256 * 192) une (96 * 128 * 160). La taille de la cible est choisie de manière à ce que la majeure partie du crâne soit capturée et, après l'avoir recadré et redimensionné, a un effet de centrage sur les images.
-
Normalisation de l'intensité
La normalisation modifie et met à l'échelle une image de sorte que les pixels de l'image aient une moyenne et une variance unitaire nulles. Cela aide le modèle à converger plus rapidement en éliminant les variations d'échelle. Ci-dessous le code pour cela.
prétraitement de classe(): def __init__(soi,df): self.data=df self.raw_index=[] self.mask_index=[] def bias_correction(soi): !mkdir bias_correction n4 = N4BiasFieldCorrection() n4.entrées.dimension = 3 n4.inputs.shrink_factor = 3 n4.inputs.n_iterations = [20, 10, 10, 5] index_corr=[] pour moi en tqdm(gamme(longueur(self.data))): n4.inputs.input_image = self.data.raw.iloc[je] n4.inputs.output_image="correction_biais/"+str(je)+'.nii.gz' index_corr.append('correction_biais/'+str(je)+'.nii.gz') res = n4.run() index_corr=['correction_biais/'+str(je)+'.nii.gz' pour i dans la plage(125)] Les données['bias_corr']=index_corr imprimer('Images corrigées du biais stockées à : correction_biais/') def resize_crop(soi): #Réduction de la taille de l'image en raison des contraintes de mémoire !mkdir redimensionné target_shape = np.array((96,128,160)) #réduire la taille de l'image de 256*256*192 à 96*128*160 nouvelle_résolution = [2,]*3 new_affine = np.zeros((4,4)) new_affine[:3,:3] = np.diag(nouvelle_résolution) # point de mise 0,0,0 au milieu du nouveau volume - cela pourrait être affiné à l'avenir new_affine[:3,3] = forme_cible*nouvelle_résolution/2.*-1 new_affine[3,3] = 1. index_raw=[] masque_index=[] #redimensionner l'image et le masque et stocker dans un dossier pour moi à portée(longueur(Les données)): downsampled_and_cropped_nii = resample_img(self.data.bias_corr.iloc[je], target_affine=new_affine, target_shape=target_shape, interpolation='le plus proche') downsampled_and_cropped_nii.to_filename('redimensionné/brut'+str(je)+'.nii.gz') self.raw_index.append('redimensionné/brut'+str(je)+'.nii.gz') downsampled_and_cropped_nii = resample_img(self.data.brain_mask.iloc[je], target_affine=new_affine, target_shape=target_shape, interpolation='le plus proche') downsampled_and_cropped_nii.to_filename('redimensionné/masquer'+str(je)+'.nii.gz') self.mask_index.append('redimensionné/masquer'+str(je)+'.nii.gz') retourner self.raw_index,self.mask_index def intensité_normalisation(soi): pour moi dans self.raw_index: image = sitk.ReadImage(je) resacleFilter = sitk.RescaleIntensityImageFilter() resacleFilter.SetOutputMaximum(255) resacleFilter.SetOutputMinimum(0) image = resacleFilter.Execute(image) sitk.WriteImage(image,je) imprimer('Normalisation effectuée. Images stockées sur: redimensionné/')
La modélisation
Maintenant que notre prétraitement est terminé, nous pouvons commencer à modéliser. Premier, nous allons faire un test de train divisé. Alors, nous utiliserons un générateur de données personnalisé pour alimenter le modèle en images d'entrée.
Regardons l'architecture du modèle.
def data_gen(soi,img_list, liste_masques, taille du lot):
'''Générateur de données personnalisé pour alimenter l'image au modèle'''
c = 0
n = [je pour je dans la gamme(longueur(img_list))] #Liste des images d'entraînement
aléatoire.shuffle(m)
tandis que (Vrai):
img = np.zéros((taille du lot, 96, 128, 160,1)).astype('flotter') #ajouter des dimensions supplémentaires car conv3d prend la taille du fichier 5
masque = np.zéros((taille du lot, 96, 128, 160,1)).astype('flotter')
pour moi à portée(c, c+taille_lot):
train_img = nib.load(img_list[m[je]]).obtenir_données()
train_img=np.expand_dims(train_img,-1)
train_mask = nib.load(liste_masques[m[je]]).obtenir_données()
train_mask=np.expand_dims(masque_train,-1)
img[je-c]=train_img
masque[je-c] = train_masque
c+=taille_lot
si(c+taille_lot>= longueur(img_list)):
c=0
aléatoire.shuffle(m)
rendement img,masque
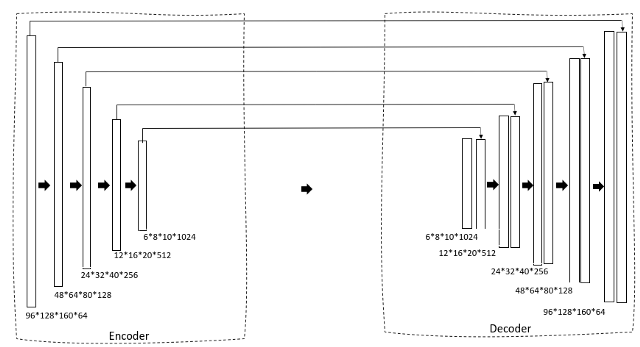
Nous utilisons un U-Net 3D comme architecture. Si vous connaissez déjà le U-Net 2D, ce sera très simple. Premier, nous avons un chemin de rétrécissement à travers un encodeur qui réduit progressivement la taille de l'image et augmente le nombre de filtres pour générer des caractéristiques de goulot d'étranglement. Ceci est ensuite transmis à un bloc décodeur qui augmente progressivement la taille afin qu'il puisse enfin générer un masque en tant que sortie prévue.
def convolutional_block(saisir, filtres=3, taille_noyau=3, batchnorm = True):
'''couche de conv suivie d'une normalisation par lots'''
x = Conv3D(filtres = filtres, taille_noyau = (taille_noyau, taille_noyau,taille_noyau),
kernel_initializer="il_normal", remplissage = 'même')(saisir)
si batchnorm:
x = LotNormalisation()(X)
x = Activation('relu')(X)
x = Conv3D(filtres = filtres, taille_noyau = (taille_noyau, taille_noyau,taille_noyau),
kernel_initializer="il_normal", remplissage = 'même')(saisir)
si batchnorm:
x = LotNormalisation()(X)
x = Activation('relu')(X)
retourner x
def resunet_opt(input_img, filtres = 64, abandon = 0.2, batchnorm = True):
"""Unet 3D résiduel"""
conv1 = bloc_convolutionnel(input_img, filtres * 1, taille_noyau = 3, batchnorm = batchnorm)
pool1 = MaxPooling3D((2, 2, 2))(conv1)
drop1 = abandon(abandonner)(piscine1)
conv2 = convolutional_block(goutte1, filtres * 2, taille_noyau = 3, batchnorm = batchnorm)
pool2 = MaxPooling3D((2, 2, 2))(conv2)
drop2 = abandon(abandonner)(piscine2)
conv3 = bloc_convolutionnel(goutte2, filtres * 4, taille_noyau = 3, batchnorm = batchnorm)
pool3 = MaxPooling3D((2, 2, 2))(conv3)
drop3 = abandon(abandonner)(piscine3)
conv4 = bloc_convolutionnel(goutte3, filtres * 8, taille_noyau = 3, batchnorm = batchnorm)
pool4 = MaxPooling3D((2, 2, 2))(conv4)
drop4 = abandon(abandonner)(piscine4)
conv5 = bloc_convolutionnel(goutte4, filtres = filtres * 16, taille_noyau = 3, batchnorm = batchnorm)
conv5 = bloc_convolutionnel(conv5, filtres = filtres * 16, taille_noyau = 3, batchnorm = batchnorm)
ups6 = Conv3DTranspose(filtres * 8, (3, 3, 3), foulées = (2, 2, 2), remplissage = 'même',activation='relu',kernel_initializer="il_normal")(conv5)
ups6 = concaténer([ups6, conv4])
ups6 = abandon(abandonner)(ups6)
conv6 = bloc_convolutionnel(ups6, filtres * 8, taille_noyau = 3, batchnorm = batchnorm)
ups7 = Conv3DTranspose(filtres * 4, (3, 3, 3), foulées = (2, 2, 2), remplissage = 'même',activation='relu',kernel_initializer="il_normal")(conv6)
ups7 = concaténer([ups7, conv3])
ups7 = abandon(abandonner)(ups7)
conv7 = bloc_convolutionnel(ups7, filtres * 4, taille_noyau = 3, batchnorm = batchnorm)
ups8 = Conv3DTranspose(filtres * 2, (3, 3, 3), foulées = (2, 2, 2), remplissage = 'même',activation='relu',kernel_initializer="il_normal")(conv7)
ups8 = concaténer([ups8, conv2])
ups8 = abandon(abandonner)(ups8)
conv8 = bloc_convolutionnel(ups8, filtres * 2, taille_noyau = 3, batchnorm = batchnorm)
ups9 = Conv3DTranspose(filtres * 1, (3, 3, 3), foulées = (2, 2, 2), remplissage = 'même',activation='relu',kernel_initializer="il_normal")(conv8)
ups9 = concaténer([ups9, conv1])
ups9 = abandon(abandonner)(ups9)
conv9 = bloc_convolutionnel(ups9, filtres * 1, taille_noyau = 3, batchnorm = batchnorm)
sorties = Conv3D(1, (1, 1, 2), activation='sigmoïde',padding='même')(conv9)
modèle = modèle(entrées =[input_img], sorties=[les sorties])
modèle de retour
Nous entraînons ensuite le modèle en utilisant l'optimiseur d'Adam et la perte de dés comme fonction de perte …
def formation(soi,époques):
im_hauteur=96
im_largeur=128
img_profondeur=160
époques=60
train_gen = data_gen(self.X_train,self.y_train, taille_bat = 4)
val_gen = data_gen(self.X_test,self.y_test, taille_bat = 4)
canaux=1
input_img = Entrée((im_hauteur, im_largeur,img_profondeur,chaînes), nom="img")
self.model = resunet_opt(input_img, filtres=16, abandon = 0,05, batchnorm=Vrai)
auto.modèle.résumé()
self.model.compile(optimiseur=Adam(lr=1e-1),perte=focal_loss,métriques=[iou_score,'précision'])
#montage du modèle
rappels=rappels = [
ModèleCheckpoint('meilleur_modèle.h5', verbeux=1, save_best_only=Vrai, save_weights_only=Faux)]
result=self.model.fit(train_gen,step_per_epoch=16,epochs=epochs,validation_data=val_gen,validation_steps=16,initial_epoch=0,callbacks=rappels)
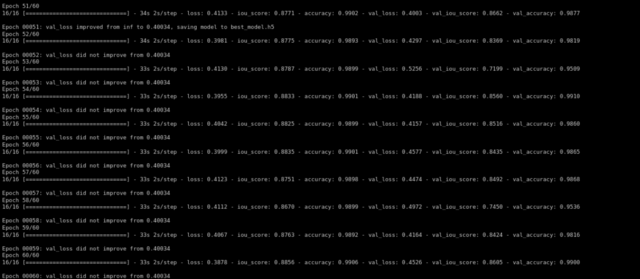
Après une formation pour 60 époques, nous avons obtenu une validation iou_score de 0.86.
Voyons comment fonctionnait notre modèle. Notre modèle va simplement prédire le masque. Pour obtenir l'image sans crâne, nous devons le superposer sur l'image Raw pour obtenir l'image sans crâne …
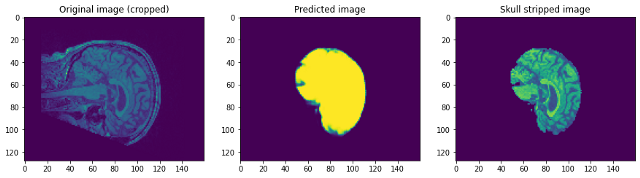
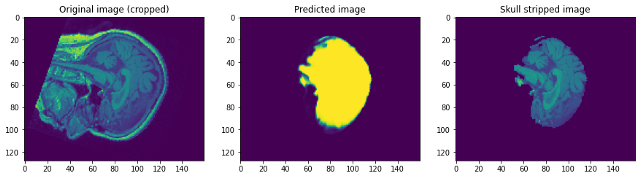

En regardant les prédictions, nous pouvons dire que bien qu'il soit capable d'identifier le cerveau et de le segmenter, pas proche de la perfection. En ce point, nous pouvons nous asseoir avec un expert du domaine pour identifier les étapes de prétraitement supplémentaires qui peuvent être effectuées pour améliorer la précision. Mais pour ce post, je vais le conclure ici. S'il vous plait, suivre lien1 Oui / O lien2 Si vous voulez en savoir plus …
conclusion:
Je suis content que tu sois allé jusqu'au bout. J'espère que cela vous aidera à démarrer avec la segmentation d'images en images 3D. Vous pouvez trouver le lien Google Colab qui contient le code. ici. N'hésitez pas à ajouter des suggestions ou des questions dans la section des commentaires. Passez une bonne journée!
Les médias présentés dans cet article sur le retrait du crâne ne sont pas la propriété de DataPeaker et sont utilisés à la discrétion de l'auteur.






