Questo articolo è stato pubblicato nell'ambito del Blogathon sulla scienza dei dati
La rimozione del cranio è uno dei passaggi preliminari per rilevare anomalie nel cervello.. È il processo di isolamento del tessuto cerebrale dal tessuto non cerebrale da un'immagine MRI di un cervello. Questo segmentazioneLa segmentazione è una tecnica di marketing chiave che comporta la divisione di un ampio mercato in gruppi più piccoli e omogenei. Questa pratica consente alle aziende di adattare le proprie strategie e i propri messaggi alle caratteristiche specifiche di ciascun segmento, migliorando così l'efficacia delle tue campagne. Il targeting può essere basato su criteri demografici, psicografico, geografico o comportamentale, facilitando una comunicazione più pertinente e personalizzata con il pubblico di destinazione.... del cerebro del cráneo es una tarea tediosa incluso para radiólogos expertos y la precisión de los resultados varía mucho de una persona a otra. Qui stiamo cercando di automatizzare il processo creando una pipeline end-to-end in cui abbiamo solo bisogno di inserire l'immagine MRI grezza e la pipeline dovrebbe generare un'immagine segmentata del cervello dopo aver eseguito la necessaria pre-elaborazione..
Quindi, Che cos'è un'immagine MRI?
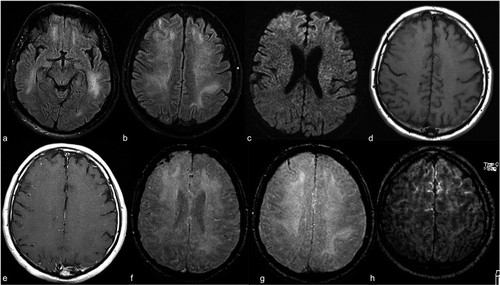
Per ottenere un'immagine MRI di un paziente, sono inseriti in un tunnel con un campo magnetico all'interno. Questo fa sì che tutti i protoni nel corpo a “allineare”, quindi il suo spin quantistico è lo stesso. Prossimo, un impulso di campo magnetico oscillante viene utilizzato per interrompere questo allineamento. Quando i protoni tornano all'equilibrio, emettere un'onda elettromagnetica. A seconda del contenuto di grassi, la composizione chimica e, cosa è più importante?, il tipo di stimolazione (vale a dire, le sequenze) usato per distruggere i protoni, si otterranno immagini diverse. Quattro sequenze comuni che si ottengono sono T1, T1 con contrasto (T1C), T2, e ISTINTO.
Sfide comuni quando si lavora con l'imaging cerebrale
-
Sfide dei dati del mondo reale
Costruire un modello e ottenere una buona precisione su un laptop jupyter è positivo. Ma la maggior parte delle volte, un modello dalle prestazioni molto buone si comporta molto male sui dati del mondo reale. Ciò accade a causa della deriva dei dati quando il modello vede dati completamente diversi da quelli per cui è stato addestrato.. Nel nostro caso, puede suceder debido a diferencias en algunos parametriIl "parametri" sono variabili o criteri che vengono utilizzati per definire, misurare o valutare un fenomeno o un sistema. In vari campi come la statistica, Informatica e Ricerca Scientifica, I parametri sono fondamentali per stabilire norme e standard che guidano l'analisi e l'interpretazione dei dati. La loro corretta selezione e gestione sono fondamentali per ottenere risultati accurati e pertinenti in qualsiasi studio o progetto.... o métodos de generación de imágenes de resonancia magnética. Ecco un Blog descrivendo alcuni errori dell'IA nel mondo reale.
Formulazione del problema
Il compito che abbiamo qui è di fornire un'immagine MRI 3D che dobbiamo identificare il cervello e segmentare il tessuto cerebrale dall'immagine completa di un cranio.. Per questo compito, avremo un tag di verità di base e, così, sarà un compito di segmentazione delle immagini supervisionato. Noi useremo perdita di dadi como nuestra Funzione di perditaLa funzione di perdita è uno strumento fondamentale nell'apprendimento automatico che quantifica la discrepanza tra le previsioni del modello e i valori effettivi. Il suo obiettivo è quello di guidare il processo di formazione minimizzando questa differenza, consentendo così al modello di apprendere in modo più efficace. Esistono diversi tipi di funzioni di perdita, come l'errore quadratico medio e l'entropia incrociata, ognuno adatto a compiti diversi e....
Dati
Diamo un'occhiata al set di dati che utilizzeremo per questo compito. Il set di dati può essere scaricato da qui.
Il repository contiene i dati di 125 partecipanti, a partire dal 21 un 45 anni, con una varietà di sintomi psichiatrici clinici e subclinici. Per ogni partecipante, il repository contiene:
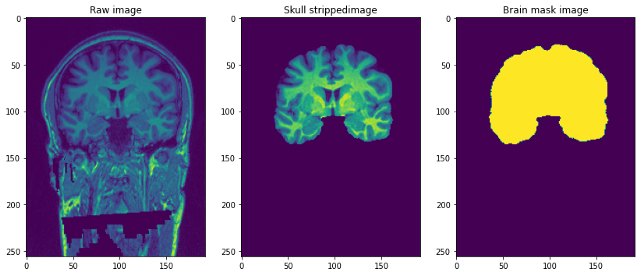
- Risonanza magnetica strutturale pesata in T1 (senza volto): questa è un'immagine MRI pesata in T1 grezza con un singolo canale.
- Maschera per il cervello: è la maschera dell'immagine del cervello o può essere chiamata la verità fondamentale?. Ottenuto usando il metodo Bestia (estrazione del cervello basata sulla segmentazione non locale) e l'applicazione di modifiche manuali da parte di esperti del dominio per rimuovere il tessuto non cerebrale.
- Immagine del teschio spogliato: questo può essere pensato come parte del cervello spogliato della precedente immagine pesata in T1. Questo è simile alla sovrapposizione di maschere su immagini reali.
Il risoluzioneIl "risoluzione" si riferisce alla capacità di prendere decisioni ferme e raggiungere gli obiettivi prefissati. In contesti personali e professionali, Implica la definizione di obiettivi chiari e lo sviluppo di un piano d'azione per raggiungerli. La risoluzione è fondamentale per la crescita personale e il successo in vari ambiti della vita, In quanto ti permette di superare gli ostacoli e mantenere la concentrazione su ciò che conta davvero.... de las imágenes es de 1 mm3 e ogni file è in formato NiFTI (.nii.gz). Un singolo punto dati assomiglia a questo …
Pre-elaborazione delle nostre immagini Raw
img=pennino.load('/content/NFBS_Dataset/A00028185/sub-A00028185_ses-NFB3_T1w.nii.gz') Stampa('Shape of image=",img.forma)

Immagina le immagini 3D sopra come se le avessimo 192 Immagini in formato 2-D 256 * 256 impilati uno sopra l'altro.
Creiamo un data frame che contenga la posizione delle immagini e le maschere corrispondenti e le immagini senza teschi.
#memorizzare l'indirizzo di 3 tipi di file
importare il sistema operativo
brain_mask=[]
cervello=[]
crudo=[]
per sottodirectory, dir, file in os.walk("/content/NFBS_Dataset'):
per file in file:
#print os.path.join(sottodirectory, file)sì
percorso file = sottodirectory + os.sep + file
if filepath.endswith(".gz"):
if '_brainmask.' in filepath:
brain_mask.append(percorso del file)
elif '_brain.' in filepath:
cervello.append(percorso del file)
altro:
raw.append(percorso del file)
Il segnale del campo di polarizzazione è un segnale a bassa frequenza molto morbido che danneggia le immagini MRI., specialmente quelli prodotti da vecchie risonanze magnetiche (Risonanza magnetica) macchine. Algoritmi di elaborazione delle immagini come la segmentazione, l'analisi della trama o la classificazione utilizzando i valori del livello di grigio dei pixel dell'immagine non produrranno risultati soddisfacenti. È necessaria una fase di pre-elaborazione per correggere il segnale del campo polarizzante prima di inviare immagini MRI danneggiate a tali algoritmi o gli algoritmi devono essere modificati.
-
Ritaglia e ridimensiona
A causa delle limitazioni computazionali dell'adattamento dell'immagine completa al modello qui, Abbiamo deciso di ridurre le dimensioni dell'immagine MRI da (256 * 256 * 192) un (96 * 128 * 160). La dimensione del bersaglio è scelta in modo tale che la maggior parte del cranio venga catturata e, dopo averlo ritagliato e ridimensionato, ha un effetto di centratura sulle immagini.
-
NormalizzazioneLa standardizzazione è un processo fondamentale in diverse discipline, che mira a stabilire norme e criteri uniformi per migliorare la qualità e l'efficienza. In contesti come l'ingegneria, Istruzione e amministrazione, La standardizzazione facilita il confronto, Interoperabilità e comprensione reciproca. Nell'attuazione degli standard, si promuove la coesione e si ottimizzano le risorse, che contribuisce allo sviluppo sostenibile e al miglioramento continuo dei processi.... de la intensidad
La normalizzazione modifica e ridimensiona un'immagine in modo che i pixel nell'immagine abbiano media zero e varianza unitaria. Ciò aiuta il modello a convergere più rapidamente eliminando la variazione di scala. Di seguito è riportato il codice per esso.
pre-elaborazione della classe(): def __init__(se stesso,df): self.data=df self.raw_index=[] self.mask_index=[] def bias_correction(se stesso): !mkdir bias_correction n4 = N4BiasFieldCorrection() n4.input.dimensione = 3 n4.inputs.shrink_factor = 3 n4.inputs.n_iterations = [20, 10, 10, 5] index_corr=[] per io in tqdm(gamma(len(self.data))): n4.inputs.input_image = self.data.raw.iloc[io] n4.inputs.output_image="correzione_bias/"+str(io)+'.nii.gz' index_corr.append('bias_correction/'+str(io)+'.nii.gz') res = n4.run() index_corr=['bias_correction/'+str(io)+'.nii.gz' for i in range(125)] dati['bias_corr']= indice_corr Stampa('Bias corrected images stored at : bias_correction/') def resize_crop(se stesso): #Ridurre le dimensioni dell'immagine a causa di vincoli di memoria !mkdir ridimensionato target_shape = np.array((96,128,160)) #riduzione delle dimensioni dell'immagine da 256*256*192 a 96*128*160 nuova_risoluzione = [2,]*3 new_affine = np.zero((4,4)) new_affine[:3,:3] = np.diag(nuova_risoluzione) # punto di messa 0,0,0 a metà del nuovo volume - questo potrebbe essere perfezionato in futuro new_affine[:3,3] = forma_obiettivo*nuova_risoluzione/2.*-1 new_affine[3,3] = 1. raw_index=[] mask_index=[] #ridimensionando sia l'immagine che la maschera e memorizzando nella cartella per io nel raggio d'azione(len(dati)): downsampled_and_cropped_nii = resample_img(self.data.bias_corr.iloc[io], target_affine=new_affine, target_shape=target_shape, interpolation='nearest') downsampled_and_cropped_nii.to_filename('resized/raw'+str(io)+'.nii.gz') self.raw_index.append('resized/raw'+str(io)+'.nii.gz') downsampled_and_cropped_nii = resample_img(self.data.brain_mask.iloc[io], target_affine=new_affine, target_shape=target_shape, interpolation='nearest') downsampled_and_cropped_nii.to_filename('resized/mask'+str(io)+'.nii.gz') self.mask_index.append('resized/mask'+str(io)+'.nii.gz') return self.raw_index,self.mask_index def intensità_normalizzazione(se stesso): per io in self.raw_index: image = sitk.ReadImage(io) resacleFilter = sitk.RescaleIntensityImageFilter() resacleFilter.SetOutputMaximum(255) resacleFilter.SetOutputMinimum(0) image = resacleFilter.Execute(Immagine) sitk.WriteImage(Immagine,io) Stampa('Normalization done. Immagini archiviate su: resized/')
modellazione
Ora che la nostra pre-elaborazione è terminata, possiamo iniziare a modellare. Primo, faremo un test del treno diviso. Dopo, useremo un generatore di dati personalizzato per alimentare le immagini di input al modello.
Diamo un'occhiata all'architettura del modello.
def data_gen(se stesso,img_list, lista_maschera, dimensione del lotto):
'''Custom data generator to feed image to model'''
c = 0
n = [io per io nel raggio d'azione(len(img_list))] #Elenco delle immagini di allenamento
random.shuffle(n)
mentre (Vero):
img = np.zero((dimensione del lotto, 96, 128, 160,1)).come tipo('float') #aggiungendo dimensioni extra poiché conv3d prende il file di dimensioni 5
maschera = np.zero((dimensione del lotto, 96, 128, 160,1)).come tipo('float')
per io nel raggio d'azione(C, c+batch_size):
train_img = pennino.load(img_list[n[io]]).get_data()
train_img=np.expand_dims(train_img,-1)
train_mask = nib.load(lista_maschera[n[io]]).get_data()
train_mask=np.expand_dims(treno_maschera,-1)
img[circuito integrato]=train_img
maschera[circuito integrato] = maschera_treno
c+=dimensione_lotto
Se(c+batch_size>= len(img_list)):
c=0
random.shuffle(n)
resa img,maschera
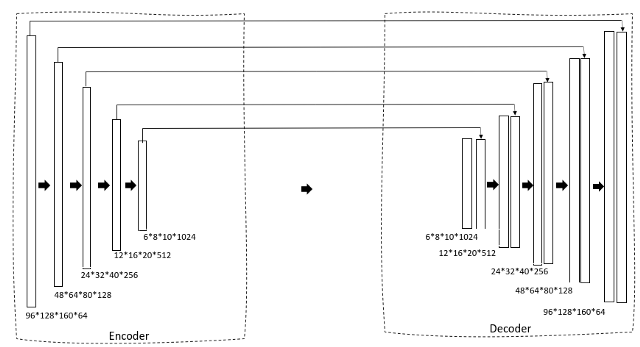
Usiamo un U-Net 3D come nostra architettura. Se hai già familiarità con U-Net 2D, sarà molto semplice. Primo, abbiamo un percorso di restringimento attraverso un encoder che riduce gradualmente la dimensione dell'immagine e aumenta il numero di filtri per generare caratteristiche di collo di bottiglia. Questo viene quindi inviato a un blocco decodificatore che espande gradualmente le dimensioni in modo che possa finalmente generare una maschera come output previsto.
def blocco_convoluzionale(ingresso, filtri=3, kernel_size=3, batchnorm = True):
'''conv layer followed by batchnormalization'''
x = Conv3D(filtri = filtri, kernel_size = (kernel_size, kernel_size,kernel_size),
kernel_initializer="lui_normale", padding = 'same')(ingresso)
se batchnorm:
x = Normalizzazione batch()(X)
x = attivazione('relu')(X)
x = Conv3D(filtri = filtri, kernel_size = (kernel_size, kernel_size,kernel_size),
kernel_initializer="lui_normale", padding = 'same')(ingresso)
se batchnorm:
x = Normalizzazione batch()(X)
x = attivazione('relu')(X)
restituire x
def resunet_opt(input_img, filtri = 64, abbandono = 0.2, batchnorm = True):
"""Unità 3D residua"""
conv1 = blocco_convoluzionale(input_img, filtri * 1, kernel_size = 3, batchnorm = batchnorm)
pool1 = MaxPooling3D((2, 2, 2))(conv1)
drop1 = Dropout(ritirarsi)(piscina1)
conv2 = blocco_convoluzionale(goccia1, filtri * 2, kernel_size = 3, batchnorm = batchnorm)
pool2 = MaxPooling3D((2, 2, 2))(conv2)
drop2 = abbandono(ritirarsi)(piscina2)
conv3 = blocco_convoluzionale(goccia2, filtri * 4, kernel_size = 3, batchnorm = batchnorm)
pool3 = MaxPooling3D((2, 2, 2))(conv3)
drop3 = Abbandono(ritirarsi)(piscina3)
conv4 = blocco_convoluzionale(goccia3, filtri * 8, kernel_size = 3, batchnorm = batchnorm)
pool4 = MaxPooling3D((2, 2, 2))(conv4)
drop4 = Abbandono(ritirarsi)(piscina4)
conv5 = blocco_convoluzionale(goccia4, filtri = filtri * 16, kernel_size = 3, batchnorm = batchnorm)
conv5 = blocco_convoluzionale(conv5, filtri = filtri * 16, kernel_size = 3, batchnorm = batchnorm)
ups6 = Conv3DTrasponi(filtri * 8, (3, 3, 3), passi = (2, 2, 2), padding = 'same',activation='relu',kernel_initializer="lui_normale")(conv5)
ups6 = concatenare([ups6, conv4])
ups6 = abbandono(ritirarsi)(ups6)
conv6 = blocco_convoluzionale(ups6, filtri * 8, kernel_size = 3, batchnorm = batchnorm)
ups7 = Conv3DTrasponi(filtri * 4, (3, 3, 3), passi = (2, 2, 2), padding = 'same',activation='relu',kernel_initializer="lui_normale")(conv6)
ups7 = concatenare([ups7, conv3])
ups7 = Abbandono(ritirarsi)(ups7)
conv7 = blocco_convoluzionale(ups7, filtri * 4, kernel_size = 3, batchnorm = batchnorm)
ups8 = Conv3DTrasponi(filtri * 2, (3, 3, 3), passi = (2, 2, 2), padding = 'same',activation='relu',kernel_initializer="lui_normale")(conv7)
ups8 = concatenare([ups8, conv2])
ups8 = Abbandono(ritirarsi)(ups8)
conv8 = blocco_convoluzionale(ups8, filtri * 2, kernel_size = 3, batchnorm = batchnorm)
ups9 = Conv3DTrasponi(filtri * 1, (3, 3, 3), passi = (2, 2, 2), padding = 'same',activation='relu',kernel_initializer="lui_normale")(conv8)
ups9 = concatenare([ups9, conv1])
ups9 = Abbandono(ritirarsi)(ups9)
conv9 = blocco_convoluzionale(ups9, filtri * 1, kernel_size = 3, batchnorm = batchnorm)
uscite = Conv3D(1, (1, 1, 2), activation='sigmoid',padding='same')(conv9)
modello = modello(ingressi=[input_img], uscite=[uscite])
modello di ritorno
Quindi addestriamo il modello utilizzando l'ottimizzatore di Adam e la perdita di dadi come funzione di perdita …
def formazione(se stesso,epoche):
im_altezza=96
im_width=128
img_depth=160
epoche=60
train_gen = data_gen(self.X_train,self.y_train, batch_size = 4)
val_gen = data_gen(self.X_test,self.y_test, batch_size = 4)
canali=1
input_img = Input((im_altezza, im_width,img_profondità,canali), nome="img")
self.model = resunet_opt(input_img, filtri=16, abbandono=0.05, batchnorm=Vero)
self.model.summary()
auto.modello.compila(ottimizzatore=Adam(lr=1e-1),perdita=perdita_focale,metriche=[iou_score,'accuracy'])
#adattando il modello
richiamate=richiamate = [
Modello Checkpoint('best_model.h5', verboso=1, save_best_only=Vero, save_weights_only=Falso)]
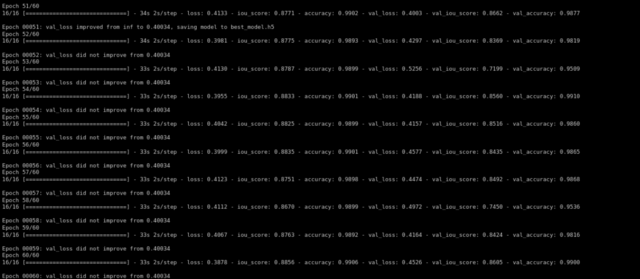
risultato=self.model.fit(train_gen,passi_per_epoca=16,epoche=epoche,validation_data=val_gen,validation_steps=16,initial_epoch=0,callback=callback)
Dopo l'allenamento per 60 epoche, abbiamo ottenuto una convalida iou_score da 0.86.
Diamo un'occhiata a come funzionava il nostro modello. Il nostro modello prevederà semplicemente la maschera. Per ottenere l'immagine senza teschio, dobbiamo sovrapporlo all'immagine Raw per ottenere l'immagine senza teschio …
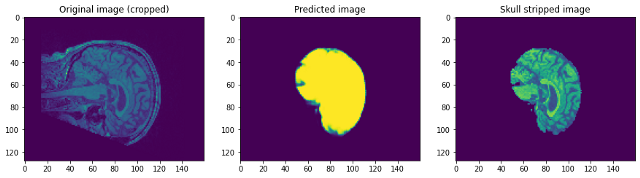
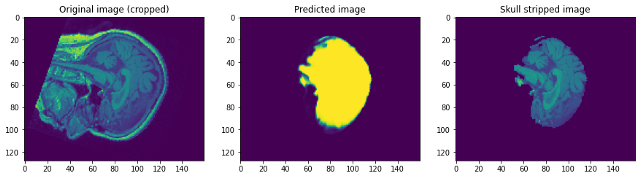

Guardando le previsioni possiamo dire che sebbene sia in grado di identificare il cervello e segmentarlo, non vicino alla perfezione. A questo punto, possiamo sederci con un esperto di dominio per identificare quali ulteriori passaggi di pre-elaborazione possono essere eseguiti per migliorare la precisione. Ma per quanto riguarda questo post, Lo concludo qui. Per favore, Seguire link1 e / oh link2 Se vuoi saperne di più …
conclusione:
Sono felice che tu sia arrivato alla fine. Spero che questo ti aiuti a iniziare con la segmentazione delle immagini in immagini 3D. Puoi trovare il link di Google Colab che contiene il codice. qui. Sentiti libero di aggiungere suggerimenti o domande nella sezione commenti. Buona giornata!
I media mostrati in questo articolo sulla rimozione del cranio non sono di proprietà di DataPeaker e vengono utilizzati a discrezione dell'autore.
Imparentato
Articoli correlati:
- Estrazione delle parole chiave | Estrazione delle parole chiave in Python
- Applicazioni di elaborazione digitale delle immagini | Elaborazione delle immagini in Python
- Classificazione delle immagini della CNN | Classificazione delle immagini utilizzando la CNN
- Elaborazione delle immagini con la CNN | Guida per principianti all'elaborazione delle immagini






