Este artigo foi publicado como parte do Data Science Blogathon
A remoção do crânio é uma das etapas preliminares para detectar anormalidades no cérebro.. É o processo de isolar tecido cerebral de tecido não cerebral a partir de uma imagem de ressonância magnética de um cérebro. Essa segmentação do cérebro a partir do crânio é uma tarefa tediosa até mesmo para radiologistas experientes, e a precisão dos resultados varia muito de pessoa para pessoa.. Aqui, estamos tentando automatizar o processo criando um pipeline de ponta a ponta em que só precisamos inserir a imagem bruta de ressonância magnética e o pipeline deve gerar uma imagem segmentada do cérebro após fazer o pré-processamento necessário..
Então, O que é uma imagem de ressonância magnética?
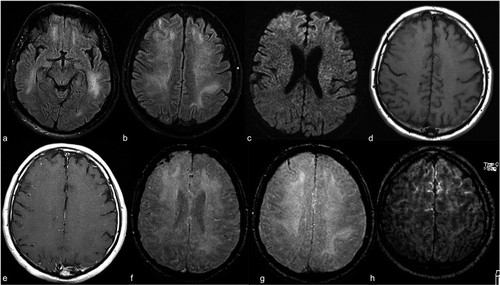
Para obter uma imagem de ressonância magnética de um paciente, são inseridos em um túnel com um campo magnético dentro. Isso faz com que todos os prótons no corpo para “alinhar”, então seu spin quântico é o mesmo. A seguir, um pulso de campo magnético oscilante é usado para interromper este alinhamento. Quando os prótons voltam ao equilíbrio, enviar uma onda eletromagnética. Dependendo do teor de gordura, a composição química e, o que é mais importante, o tipo de estimulação (quer dizer, as sequências) usado para interromper prótons, imagens diferentes serão obtidas. Quatro sequências comuns que são obtidas são T1, T1 com contraste (T1C), T2, e INSTINTO.
Desafios comuns ao trabalhar com imagens cerebrais
-
Desafios de dados do mundo real
Construir um modelo e obter boa precisão em um laptop Jupyter é bom. Mas na maioria das vezes, um modelo de muito bom desempenho tem um desempenho muito ruim em dados do mundo real. Isso acontece devido ao desvio de dados, quando o modelo vê dados completamente diferentes do que foi treinado para fazer.. No nosso caso, pode acontecer devido a diferenças em alguns parâmetros ou métodos de imagem por ressonância magnética. Aqui está um Blog descrevendo algumas falhas de IA do mundo real.
Formulação de problema
A tarefa que temos aqui é fornecer uma imagem 3D de ressonância magnética que temos para identificar o cérebro e segmentar o tecido cerebral a partir da imagem completa de um crânio.. Para esta tarefa, teremos uma etiqueta de verdade básica e, portanto, será uma tarefa de segmentação de imagem supervisionada. Nós vamos usar perda de dados como nossa função de perda.
Dados
Vamos dar uma olhada no conjunto de dados que usaremos para esta tarefa. O conjunto de dados pode ser baixado de aqui.
O repositório contém dados de 125 participantes, a partir de 21 uma 45 anos, com uma variedade de sintomas psiquiátricos clínicos e subclínicos. Para cada participante, o repositório contém:
- Imagem de ressonância magnética ponderada em T1 estrutural (sem rosto): esta é uma imagem de ressonância magnética ponderada em T1 com canal único.
- Máscara cerebral: é a máscara da imagem do cérebro ou pode ser chamada de verdade fundamental. Obtido usando o método Besta (extração do cérebro com base na segmentação não local) e aplicação de edições manuais por especialistas de domínio para remover tecido não cerebral.
- Imagem de crânio despojado: isso pode ser pensado como parte do cérebro despojado da imagem anterior ponderada em T1. Isso é semelhante a sobrepor máscaras em imagens reais.
A resolução das imagens é 1 mm3 e cada arquivo está no formato NiFTI (.nii.gz). Um único ponto de dados se parece com isto …
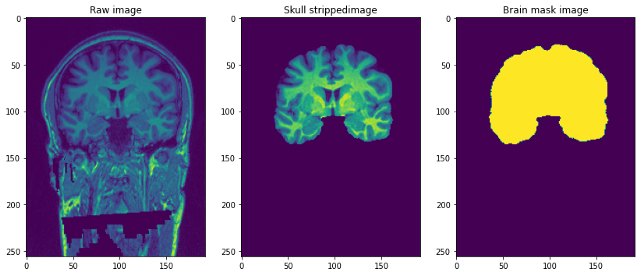
Pré-processamento de nossas imagens Raw
img = nib.load('/content/NFBS_Dataset/A00028185/sub-A00028185_ses-NFB3_T1w.nii.gz')
imprimir('Forma da imagem =",img.shape)

Imagine imagens 3-D acima como se tivéssemos 192 Imagens de tamanho 2-D 256 * 256 empilhados um em cima do outro.
Vamos criar um quadro de dados que contém a localização das imagens e as máscaras correspondentes e as imagens sem caveiras.
#armazenando o endereço de 3 tipos de arquivos
importar os
brain_mask =[]
cérebro =[]
cru =[]
para subdir, dirs, arquivos em os.walk("/content / NFBS_Dataset '):
para arquivo em arquivos:
#imprimir os.path.join(subdiretório, Arquivo)y
filepath = subdir + os.sep + Arquivo
if filepath.endswith(".gz"):
if '_brainmask.' no caminho do arquivo:
brain_mask.append(caminho de arquivo)
elif '_brain.' no caminho do arquivo:
brain.append(caminho de arquivo)
outro:
raw.append(caminho de arquivo)
O sinal do campo de polarização é um sinal de baixa frequência muito suave que corrompe as imagens de ressonância magnética., especialmente aqueles produzidos por ressonâncias magnéticas antigas (Imagem de ressonância magnética) máquinas. Algoritmos de processamento de imagem, como segmentação, análise de textura ou classificação usando valores de nível de cinza de pixel de imagem não produzirá resultados satisfatórios. Uma etapa de pré-processamento é necessária para corrigir o sinal de campo de polarização antes de enviar imagens de MRI corrompidas para tais algoritmos ou os algoritmos devem ser modificados.
-
Cortar e redimensionar
Devido às limitações computacionais de ajustar a imagem completa ao modelo aqui, Decidimos reduzir o tamanho da imagem de ressonância magnética de (256 * 256 * 192) uma (96 * 128 * 160). O tamanho do alvo é escolhido de forma que a maior parte do crânio seja capturada e, depois de cortar e redimensionar, tem um efeito de centralização nas imagens.
-
Normalização de intensidade
A normalização muda e dimensiona uma imagem para que os pixels na imagem tenham média zero e variância unitária. Isso ajuda o modelo a convergir mais rápido, eliminando a variação de escala. Abaixo está o código para isso.
pré-processamento de classe(): def __init__(auto,df): self.data = df self.raw_index =[] self.mask_index =[] def bias_correction(auto): !mkdir bias_correction n4 = N4BiasFieldCorrection() n4.inputs.dimension = 3 n4.inputs.shrink_factor = 3 n4.inputs.n_iterations = [20, 10, 10, 5] index_corr =[] para eu no tqdm(faixa(len(self.data))): n4.inputs.input_image = self.data.raw.iloc[eu] n4.inputs.output_image ="bias_correction /"+str(eu)+'.nii.gz' index_corr.append('bias_correction /' + str(eu)+'.nii.gz') res = n4.run() index_corr =['bias_correction /' + str(eu)+'.nii.gz' para i no intervalo(125)] dados['bias_corr']= index_corr imprimir(Imagens corrigidas de polarização armazenadas em : bias_correction / ') def resize_crop(auto): #Reduzindo o tamanho da imagem devido a restrições de memória !mkdir redimensionado target_shape = np.array((96,128,160)) #reduzindo o tamanho da imagem de 256*256*192 para 96*128*160 nova_resolução = [2,]*3 new_affine = np.zeros((4,4)) new_affine[:3,:3] = np.diag(nova_resolução) # ponto de colocação 0,0,0 no meio do novo volume - isso poderia ser refinado no futuro new_affine[:3,3] = target_shape * new_resolution / 2. * - 1 new_affine[3,3] = 1. raw_index =[] mask_index =[] #redimensionando a imagem e a máscara e armazenando na pasta para eu no alcance(len(dados)): downsampled_and_cropped_nii = resample_img(self.data.bias_corr.iloc[eu], target_affine = new_affine, target_shape = target_shape, interpolação = 'mais próximo') downsampled_and_cropped_nii.to_filename('redimensionado / bruto' + str(eu)+'.nii.gz') self.raw_index.append('redimensionado / bruto' + str(eu)+'.nii.gz') downsampled_and_cropped_nii = resample_img(self.data.brain_mask.iloc[eu], target_affine = new_affine, target_shape = target_shape, interpolação = 'mais próximo') downsampled_and_cropped_nii.to_filename('redimensionado / máscara' + str(eu)+'.nii.gz') self.mask_index.append('redimensionado / máscara' + str(eu)+'.nii.gz') return self.raw_index,self.mask_index def Intensity_normalization(auto): para i em self.raw_index: image = sitk.ReadImage(eu) resacleFilter = sitk.RescaleIntensityImageFilter() resacleFilter.SetOutputMaximum(255) resacleFilter.SetOutputMinimum(0) image = resacleFilter.Execute(imagem) sitk.WriteImage(imagem,eu) imprimir('Normalização feita. Imagens armazenadas em: redimensionado / ')
Modelagem
Agora que nosso pré-processamento está feito, podemos começar a modelar. Primeiro, vamos fazer um teste de trem dividido. Mais tarde, vamos usar um gerador de dados personalizados para alimentar as imagens de entrada para o modelo.
Vamos dar uma olhada na arquitetura do modelo.
def data_gen(auto,img_list, mask_list, tamanho do batch):
'' 'Gerador de dados personalizados para alimentar a imagem do modelo' ''
c = 0
n = [i para i no alcance(len(img_list))] #Lista de imagens de treinamento
random.shuffle(n)
enquanto (Verdade):
img = np.zeros((tamanho do batch, 96, 128, 160,1)).astype('flutuador') #adicionar dimensões extras à medida que o conv3d pega o arquivo de tamanho 5
mask = np.zeros((tamanho do batch, 96, 128, 160,1)).astype('flutuador')
para eu no alcance(c, c + batch_size):
train_img = nib.load(img_list[n[eu]]).Obter dados()
train_img = np.expand_dims(train_img,-1)
train_mask = nib.load(mask_list[n[eu]]).Obter dados()
train_mask = np.expand_dims(train_mask,-1)
img[i-c]= train_img
mascarar[i-c] = train_mask
c + = batch_size
E se(c + batch_size>= len(img_list)):
c = 0
random.shuffle(n)
rendimento img,mascarar
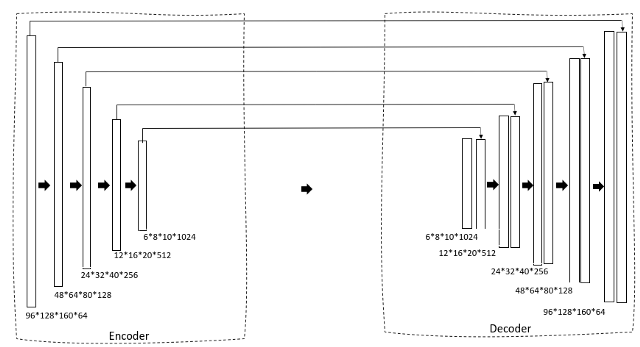
Estamos usando um U-Net 3D como nossa arquitetura. Se você já está familiarizado com o U-Net 2D, isso vai ser muito simples. Primeiro, temos um caminho de redução através de um codificador que reduz gradualmente o tamanho da imagem e aumenta o número de filtros para gerar características de gargalo. Isso é então alimentado para um bloco decodificador que gradualmente expande o tamanho para que possa finalmente gerar uma máscara como a saída pretendida.
def convolutional_block(entrada, filtros = 3, kernel_size = 3, batchnorm = True):
'' 'camada conv seguida por normalização em lote' ''
x = Conv3D(filtros = filtros, kernel_size = (kernel_size, kernel_size,kernel_size),
kernel_initializer ="he_normal", preenchimento = 'mesmo')(entrada)
se batchnorm:
x = BatchNormalization()(x)
x = Ativação('relu')(x)
x = Conv3D(filtros = filtros, kernel_size = (kernel_size, kernel_size,kernel_size),
kernel_initializer ="he_normal", preenchimento = 'mesmo')(entrada)
se batchnorm:
x = BatchNormalization()(x)
x = Ativação('relu')(x)
retornar x
def resunet_opt(input_img, filtros = 64, dropout = 0.2, batchnorm = True):
"""Residual 3D Unet"""
conv1 = convolutional_block(input_img, filtros * 1, kernel_size = 3, batchnorm = batchnorm)
pool1 = MaxPooling3D((2, 2, 2))(conv1)
drop1 = Dropout(cair fora)(pool1)
conv2 = convolutional_block(drop1, filtros * 2, kernel_size = 3, batchnorm = batchnorm)
pool2 = MaxPooling3D((2, 2, 2))(conv2)
drop2 = Dropout(cair fora)(pool2)
conv3 = convolutional_block(drop2, filtros * 4, kernel_size = 3, batchnorm = batchnorm)
pool3 = MaxPooling3D((2, 2, 2))(conv3)
drop3 = desistência(cair fora)(pool3)
conv4 = convolutional_block(drop3, filtros * 8, kernel_size = 3, batchnorm = batchnorm)
pool4 = MaxPooling3D((2, 2, 2))(conv4)
drop4 = desistência(cair fora)(pool4)
conv5 = convolutional_block(drop4, filtros = filtros * 16, kernel_size = 3, batchnorm = batchnorm)
conv5 = convolutional_block(conv5, filtros = filtros * 16, kernel_size = 3, batchnorm = batchnorm)
ups6 = Conv3DTranspose(filtros * 8, (3, 3, 3), passos largos = (2, 2, 2), preenchimento = 'mesmo',ativação = 'reler',kernel_initializer ="he_normal")(conv5)
ups6 = concatenar([ups6, conv4])
ups6 = Desistência(cair fora)(ups6)
conv6 = convolutional_block(ups6, filtros * 8, kernel_size = 3, batchnorm = batchnorm)
ups7 = Conv3DTranspose(filtros * 4, (3, 3, 3), passos largos = (2, 2, 2), preenchimento = 'mesmo',ativação = 'reler',kernel_initializer ="he_normal")(conv6)
ups7 = concatenar([ups7, conv3])
ups7 = Desistência(cair fora)(ups7)
conv7 = convolutional_block(ups7, filtros * 4, kernel_size = 3, batchnorm = batchnorm)
ups8 = Conv3DTranspose(filtros * 2, (3, 3, 3), passos largos = (2, 2, 2), preenchimento = 'mesmo',ativação = 'reler',kernel_initializer ="he_normal")(conv7)
ups8 = concatenar([ups8, conv2])
ups8 = Desistência(cair fora)(ups8)
conv8 = convolutional_block(ups8, filtros * 2, kernel_size = 3, batchnorm = batchnorm)
ups9 = Conv3DTranspose(filtros * 1, (3, 3, 3), passos largos = (2, 2, 2), preenchimento = 'mesmo',ativação = 'reler',kernel_initializer ="he_normal")(conv8)
ups9 = concatenar([ups9, conv1])
ups9 = Desistência(cair fora)(ups9)
conv9 = convolutional_block(ups9, filtros * 1, kernel_size = 3, batchnorm = batchnorm)
saídas = Conv3D(1, (1, 1, 2), ativação = 'sigmóide',preenchimento = 'mesmo')(conv9)
model = model(entradas =[input_img], saídas =[saídas])
modelo de retorno
Em seguida, treinamos o modelo usando o otimizador de Adam e a perda de dados como nossa função de perda …
treinamento de def(auto,épocas):
im_height = 96
largura_im = 128
img_depth = 160
épocas = 60
train_gen = data_gen(self.X_train,self.y_train, batch_size = 4)
val_gen = data_gen(self.X_test,self.y_test, batch_size = 4)
canais = 1
input_img = Input((im_height, im_width,img_depth,canais), nome ="img")
self.model = resunet_opt(input_img, filtros = 16, abandono = 0,05, batchnorm = True)
self.model.summary()
self.model.compile(otimizador = Adam(lr = 1e-1),perda = perda_focal,metrics =[iou_score,'precisão'])
#ajustando o modelo
callbacks = callbacks = [
ModelCheckpoint('best_model.h5', verbose = 1, save_best_only = True, save_weights_only = False)]
resultado = self.model.fit(train_gen,steps_per_epoch = 16, epochs = epochs,validação_data = val_gen,validation_steps = 16, initial_epoch = 0, callbacks = callbacks)
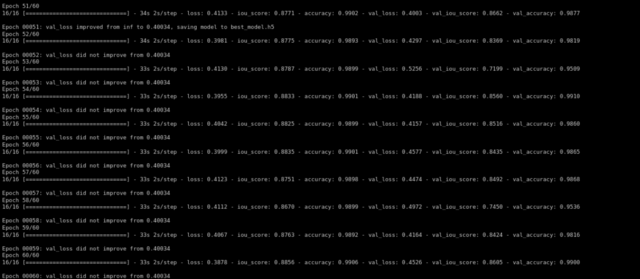
Depois de treinar para 60 épocas, temos uma validação iou_score de 0.86.
Vamos dar uma olhada em como nosso modelo funcionava. Nosso modelo irá simplesmente prever a máscara. Para obter a imagem sem caveira, precisamos sobrepô-lo na imagem Raw para obter a imagem sem caveira …
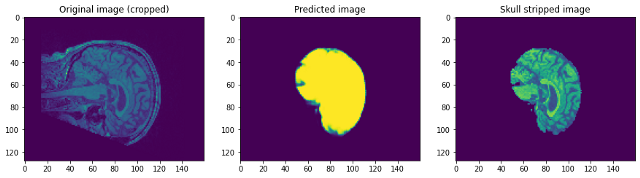
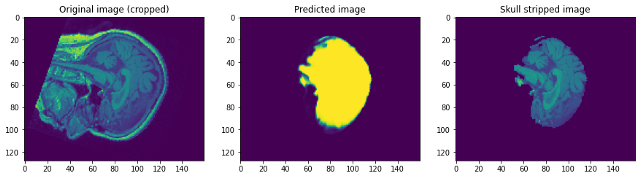

Olhando para as previsões podemos dizer que embora seja capaz de identificar o cérebro e segmentá-lo, não perto da perfeição. Neste ponto, podemos conversar com um especialista de domínio para identificar quais etapas adicionais de pré-processamento podem ser realizadas para melhorar a precisão. Mas quanto a este post, Vou concluir aqui. Por favor, siga link1 e / o link2 Se você quiser saber mais …
conclusão:
Estou feliz que você tenha chegado ao fim. Espero que isso ajude você a começar com a segmentação de imagens em imagens 3D. Você pode encontrar o link do Google Colab que contém o código. aqui. Sinta-se à vontade para adicionar sugestões ou perguntas na seção de comentários. Que tenha um lindo dia!
A mídia mostrada neste artigo de remoção de crânio não é propriedade da DataPeaker e é usada a critério do autor.






