Dieser Artikel wurde im Rahmen der Data Science Blogathon
Die Entfernung des Schädels ist einer der ersten Schritte auf dem Weg zur Erkennung von Anomalien im Gehirn.. Es ist der Prozess der Isolierung von Hirngewebe von Nicht-Gehirngewebe aus einem MRT-Bild eines Gehirns. Diese Segmentierung des Gehirns vom Schädel ist selbst für erfahrene Radiologen eine mühsame Aufgabe und die Genauigkeit der Ergebnisse variiert stark von Person zu Person.. Hier versuchen wir, den Prozess zu automatisieren, indem wir eine End-to-End-Pipeline erstellen, bei der wir nur das MRT-Rohbild eingeben müssen und die Pipeline nach der erforderlichen Vorverarbeitung ein segmentiertes Bild des Gehirns generieren soll..
Dann, Was ist ein MRT-Bild??
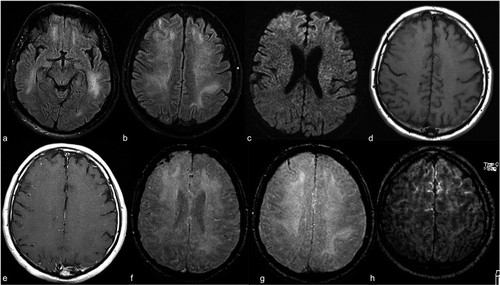
Um ein MRT-Bild eines Patienten zu erhalten, werden in einen Tunnel mit einem Magnetfeld im Inneren eingeführt. Dadurch werden alle Protonen im Körper “ausrichten”, also ist sein Quantenspin derselbe. Dann, ein oszillierender Magnetfeldimpuls wird verwendet, um diese Ausrichtung zu stören. Wenn die Protonen ins Gleichgewicht zurückkehren, eine elektromagnetische Welle aussenden. Je nach Fettgehalt, die chemische Zusammensetzung und, Was ist wichtiger, die Art der Stimulation (nämlich, die sequenzen) verwendet, um Protonen zu zerstören, verschiedene Bilder werden erhalten. Vier häufige Sequenzen, die erhalten werden, sind T1, T1 mit Kontrast (T1C), T2, Ja INSTINKT.
Häufige Herausforderungen bei der Arbeit mit der Bildgebung des Gehirns
-
Herausforderungen realer Daten
Ein Modell zu bauen und eine gute Genauigkeit auf einem Jupyter-Laptop zu erreichen, ist gut. Aber die meiste Zeit, ein Modell mit sehr guter Leistung schneidet bei realen Daten sehr schlecht ab. Dies geschieht aufgrund von Datendrift, wenn das Modell völlig andere Daten sieht, als es trainiert wurde.. In unserem Fall, es kann aufgrund von Unterschieden in einigen Parametern oder Magnetresonanztomographie-Methoden vorkommen. Hier ist ein Blog Beschreibung einiger realer KI-Fehler.
Problem Formulierung
Die Aufgabe, die wir hier haben, besteht darin, ein 3D-MRT-Bild zu erstellen, mit dem wir das Gehirn identifizieren und das Gehirngewebe aus dem Vollbild eines Schädels segmentieren müssen.. Für diese Aufgabe, wir werden ein grundlegendes Wahrheits-Tag haben und, Daher, wird eine überwachte Bildsegmentierungsaufgabe sein. Wir werden verwenden Verlust von Würfeln als unsere Verlustfunktion.
Daten
Werfen wir einen Blick auf den Datensatz, den wir für diese Aufgabe verwenden werden. Der Datensatz kann heruntergeladen werden unter hier.
Das Repository enthält Daten von 125 Teilnehmer, von 21 ein 45 Jahre, mit einer Vielzahl von klinischen und subklinischen psychiatrischen Symptomen. Für jeden Teilnehmer, das Repository enthält:
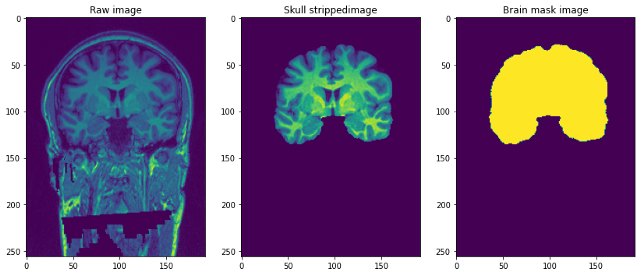
- Strukturelle T1-gewichtete Magnetresonanztomographie (gesichtslos): Dies ist ein T1-gewichtetes MRT-Rohbild mit einem einzigen Kanal.
- Gehirnmaske: ist die Bildmaske des Gehirns oder kann man sie die fundamentale Wahrheit nennen. Erhalten mit der Beast-Methode (Gehirnextraktion basierend auf nicht-lokaler Segmentierung) und Anwenden manueller Bearbeitungen durch Domänenexperten, um Nicht-Hirn-Gewebe zu entfernen.
- Abgestreiftes Schädelbild: dies kann man sich als Teil des Gehirns vorstellen, dem das vorherige T1-gewichtete Bild entzogen wurde. Dies ist vergleichbar mit dem Überlagern von Masken auf realen Bildern.
Die Auflösung der Bilder ist 1 mm3 und jede Datei ist im NiFTI-Format (.nii.gz). Ein einzelner Datenpunkt sieht so aus …
Vorverarbeitung unserer Raw-Bilder
img=nib.load('/content/NFBS_Dataset/A00028185/sub-A00028185_ses-NFB3_T1w.nii.gz')
drucken('Bildform=",img.shape)

Stellen Sie sich die 3D-Bilder oben vor, als hätten wir es getan 192 Bilder in 2D-Größe 256 * 256 übereinander gestapelt.
Lassen Sie uns einen Datenrahmen erstellen, der die Position der Bilder und der entsprechenden Masken und der Bilder ohne Schädel enthält.
#Speichern der Adresse von 3 Dateitypen
Importieren von OS
brain_mask=[]
Gehirn=[]
roh=[]
für Unterverzeichnis, dirs, Dateien in os.walk("/Inhalt/NFBS_Dataset'):
für Datei in Dateien:
#os.path.join drucken(Unterverzeichnis, Datei)ja
Dateipfad = Unterverzeichnis + os.sep + Datei
if filepath.endswith(".gz"):
if '_Gehirnmaske.' im Dateipfad:
brain_mask.append(Dateipfad)
elif '_hirn.' im Dateipfad:
Gehirn.anhängen(Dateipfad)
anders:
raw.anhängen(Dateipfad)
Das Polarisationsfeldsignal ist ein sehr weiches, niederfrequentes Signal, das MRT-Bilder verfälscht., insbesondere solche, die von alten MRTs erzeugt werden (Magnetresonanztomographie) Maschinen. Bildverarbeitungsalgorithmen wie Segmentierung, Texturanalyse oder Klassifizierung anhand von Bildpixel-Graustufenwerten führt nicht zu zufriedenstellenden Ergebnissen. Ein Vorverarbeitungsschritt ist erforderlich, um das Polarisationsfeldsignal zu korrigieren, bevor beschädigte MRT-Bilder an solche Algorithmen gesendet werden, oder die Algorithmen müssen modifiziert werden.
-
Zuschneiden und Größe ändern
Aufgrund von rechnerischen Einschränkungen bei der Anpassung des vollständigen Bildes an das Modell hier, Wir haben uns entschieden, die Größe des MRT-Bildes von (256 * 256 * 192) ein (96 * 128 * 160). Die Größe des Ziels ist so gewählt, dass der größte Teil des Schädels erfasst wird und, nach dem Zuschneiden und Ändern der Größe, wirkt zentrierend auf Bilder.
-
Intensitätsnormalisierung
Die Normalisierung ändert und skaliert ein Bild, sodass die Pixel im Bild null Mittelwert und Einheitsvarianz aufweisen. Dies hilft dem Modell, schneller zu konvergieren, indem Skalenabweichungen eliminiert werden. Unten ist der Code dafür.
Klassenvorverarbeitung(): def __init__(selbst,df): self.data=df self.raw_index=[] self.mask_index=[] def Bias_Korrektur(selbst): !mkdir bias_korrektur n4 = N4BiasFieldCorrection() n4.Eingänge.Maß = 3 n4.inputs.shrink_factor = 3 n4.inputs.n_iterations = [20, 10, 10, 5] index_corr=[] für ich in tqdm(Bereich(len(self.data))): n4.inputs.input_image = self.data.raw.iloc[ich] n4.inputs.output_image="Voreingenommenheit_Korrektur/"+str(ich)+'.nii.gz' index_korr.anhängen('bias_correction/'+str(ich)+'.nii.gz') res = n4.run() index_corr=['bias_correction/'+str(ich)+'.nii.gz' für i in Reichweite(125)] Daten['bias_korr']=index_korr drucken('Bias-korrigierte Bilder gespeichert unter : Voreingenommenheit_Korrektur/') def resize_crop(selbst): #Reduzieren der Bildgröße aufgrund von Speicherbeschränkungen !mkdir in der Größe geändert target_shape = np.array((96,128,160)) #Verkleinerung des Bildes von 256*256*192 zu 96*128*160 neue_auflösung = [2,]*3 new_affine = np.null((4,4)) neu_affin[:3,:3] = np.diag(neue_auflösung) # Punkt setzen 0,0,0 mitten im neuen Band - das könnte in Zukunft verfeinert werden neu_affin[:3,3] = Zielform*neue_Auflösung/2.*-1 neu_affin[3,3] = 1. raw_index=[] mask_index=[] #Größe von Bild und Maske ändern und im Ordner speichern für mich in Reichweite(len(Daten)): downsampled_and_cropped_nii = resample_img(self.data.bias_corr.iloc[ich], target_affine=new_affine, target_shape=target_shape, interpolation='nächste') downsampled_and_cropped_nii.to_filename('Größe geändert/roh'+str(ich)+'.nii.gz') self.raw_index.append('Größe geändert/roh'+str(ich)+'.nii.gz') downsampled_and_cropped_nii = resample_img(self.data.brain_mask.iloc[ich], target_affine=new_affine, target_shape=target_shape, interpolation='nächste') downsampled_and_cropped_nii.to_filename('Größe/Maske'+Str(ich)+'.nii.gz') self.mask_index.append('Größe/Maske'+Str(ich)+'.nii.gz') Rückgabe self.raw_index,self.mask_index def Intensität_Normalisierung(selbst): für i in self.raw_index: image = sitk.ReadImage(ich) resacleFilter = sitk.RescaleIntensityImageFilter() resacleFilter.SetOutputMaximum(255) resacleFilter.SetOutputMinimum(0) image = resacleFilter.Execute(Bild) sitk.WriteImage(Bild,ich) drucken('Normalisierung abgeschlossen. Bilder gespeichert unter: verkleinert/')
Modellieren
Nun, da unsere Vorverarbeitung abgeschlossen ist, wir können anfangen zu modellieren. Zuerst, Wir machen einen Split-Train-Test. Später, Wir werden einen benutzerdefinierten Datengenerator verwenden, um die Eingabebilder in das Modell einzuspeisen.
Werfen wir einen Blick auf die Architektur des Modells.
def data_gen(selbst,img_list, mask_list, batch_size):
'''Benutzerdefinierter Datengenerator zum Einspeisen von Bildern in das Modell'''
c = 0
n = [i für i in Reichweite(len(img_list))] #Liste der Trainingsbilder
random.shuffle(n)
während (Wahr):
img = np.null((batch_size, 96, 128, 160,1)).astyp('schweben') #Hinzufügen zusätzlicher Dimensionen, da conv3d die Dateigröße nimmt 5
Maske = np.null((batch_size, 96, 128, 160,1)).astyp('schweben')
für mich in Reichweite(C, c+batch_größe):
train_img = nib.load(img_list[n[ich]]).Daten bekommen()
train_img=np.expand_dims(train_img,-1)
train_mask = nib.load(mask_list[n[ich]]).Daten bekommen()
train_mask=np.expand_dims(train_mask,-1)
img[NS]=train_img
Maske[NS] = train_mask
c+=batch_size
wenn(c+batch_größe>= len(img_list)):
c=0
random.shuffle(n)
Ertrag img,Maske
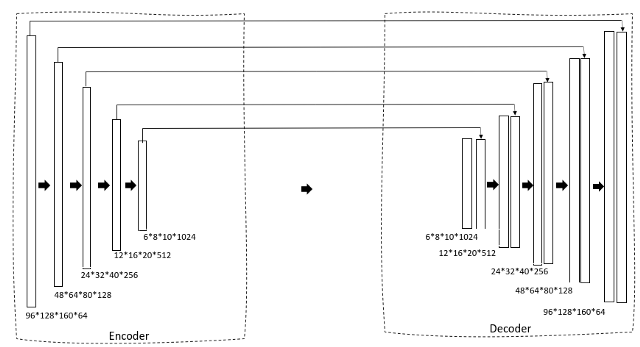
Wir verwenden ein U-Net 3D als unsere Architektur. Wenn Sie mit dem U-Net 2D bereits vertraut sind, das wird ganz einfach. Zuerst, Wir haben einen Schrumpfpfad durch einen Encoder, der die Bildgröße allmählich reduziert und die Anzahl der Filter erhöht, um Engpasseigenschaften zu erzeugen. Dieser wird dann einem Decoderblock zugeführt, der die Größe nach und nach erweitert, um schließlich eine Maske als beabsichtigte Ausgabe zu erzeugen.
def Faltungsblock(Eingang, Filter=3, kernel_size=3, Batchnorm = True):
'''conv-Layer gefolgt von Batchnormalisierung'''
x = Conv3D(Filter = Filter, Kernelgröße = (Kernelgröße, Kernelgröße,Kernelgröße),
kernel_initializer="er_normal", Polsterung = 'gleich')(Eingang)
wenn Batchnorm:
x = BatchNormalisierung()(x)
x = Aktivierung('relu')(x)
x = Conv3D(Filter = Filter, Kernelgröße = (Kernelgröße, Kernelgröße,Kernelgröße),
kernel_initializer="er_normal", Polsterung = 'gleich')(Eingang)
wenn Batchnorm:
x = BatchNormalisierung()(x)
x = Aktivierung('relu')(x)
Rückgabe x
def resunet_opt(input_img, Filter = 64, Ausfall = 0.2, Batchnorm = True):
"""Rest 3D Unet"""
conv1 = Faltungsblock(input_img, Filter * 1, Kernelgröße = 3, Batchnorm = Batchnorm)
pool1 = MaxPooling3D((2, 2, 2))(conv1)
drop1 = Ausfall(aussteigen)(Pool1)
conv2 = Faltungsblock(drop1, Filter * 2, Kernelgröße = 3, Batchnorm = Batchnorm)
pool2 = MaxPooling3D((2, 2, 2))(conv2)
drop2 = Ausfall(aussteigen)(Pool2)
conv3 = Faltungsblock(drop2, Filter * 4, Kernelgröße = 3, Batchnorm = Batchnorm)
pool3 = MaxPooling3D((2, 2, 2))(conv3)
drop3 = Ausfall(aussteigen)(Pool3)
conv4 = Faltungsblock(drop3, Filter * 8, Kernelgröße = 3, Batchnorm = Batchnorm)
pool4 = MaxPooling3D((2, 2, 2))(conv4)
drop4 = Ausfall(aussteigen)(Pool4)
conv5 = Faltungsblock(drop4, Filter = Filter * 16, Kernelgröße = 3, Batchnorm = Batchnorm)
conv5 = Faltungsblock(conv5, Filter = Filter * 16, Kernelgröße = 3, Batchnorm = Batchnorm)
ups6 = Conv3DTranspose(Filter * 8, (3, 3, 3), Schritte = (2, 2, 2), Polsterung = 'gleich',Aktivierung = 'neu lesen',kernel_initializer="er_normal")(conv5)
ups6 = verketten([ups6, conv4])
ups6 = Aussteiger(aussteigen)(ups6)
conv6 = Faltungsblock(ups6, Filter * 8, Kernelgröße = 3, Batchnorm = Batchnorm)
ups7 = Conv3DTranspose(Filter * 4, (3, 3, 3), Schritte = (2, 2, 2), Polsterung = 'gleich',Aktivierung = 'neu lesen',kernel_initializer="er_normal")(conv6)
ups7 = verketten([ups7, conv3])
ups7 = Aussteiger(aussteigen)(ups7)
conv7 = Faltungsblock(ups7, Filter * 4, Kernelgröße = 3, Batchnorm = Batchnorm)
ups8 = Conv3DTranspose(Filter * 2, (3, 3, 3), Schritte = (2, 2, 2), Polsterung = 'gleich',Aktivierung = 'neu lesen',kernel_initializer="er_normal")(conv7)
ups8 = verketten([ups8, conv2])
ups8 = Aussteiger(aussteigen)(ups8)
conv8 = Faltungsblock(ups8, Filter * 2, Kernelgröße = 3, Batchnorm = Batchnorm)
ups9 = Conv3DTranspose(Filter * 1, (3, 3, 3), Schritte = (2, 2, 2), Polsterung = 'gleich',Aktivierung = 'neu lesen',kernel_initializer="er_normal")(conv8)
ups9 = verketten([ups9, conv1])
ups9 = Aussteiger(aussteigen)(ups9)
conv9 = Faltungsblock(ups9, Filter * 1, Kernelgröße = 3, Batchnorm = Batchnorm)
Ausgänge = Conv3D(1, (1, 1, 2), Aktivierung='Sigmoid',padding='gleich')(conv9)
Modell = Modell(Eingänge=[input_img], Ausgänge=[Ausgänge])
Rückgabemodell
Dann trainieren wir das Modell mit Adams Optimierer und Würfelverlust als Verlustfunktion …
Def-Training(selbst,Epochen):
im_höhe=96
im_width=128
img_depth=160
Epochen=60
train_gen = data_gen(self.X_train,self.y_train, batch_size = 4)
val_gen = data_gen(self.X_test,self.y_test, batch_size = 4)
Kanäle=1
input_img = Eingabe((im_höhe, im_width,img_depth,Kanäle), name="img")
self.model = resunet_opt(input_img, Filter=16, Ausfall = 0,05, Batchnorm=Wahr)
Selbstmodell.Zusammenfassung()
self.model.compile(Optimierer=Adam(lr=1e-1),Verlust=Focal_Loss,Metriken=[iou_score,'Richtigkeit'])
#passend zum Modell
Rückrufe = Rückrufe = [
ModellCheckpoint('best_model.h5', ausführlich=1, save_best_only=Wahr, save_weights_only=Falsch)]
result=self.model.fit(train_gen,steps_per_epoch=16,epochs=epochs,validation_data=val_gen,validierungsschritte=16,initial_epoch=0,callbacks=callbacks)
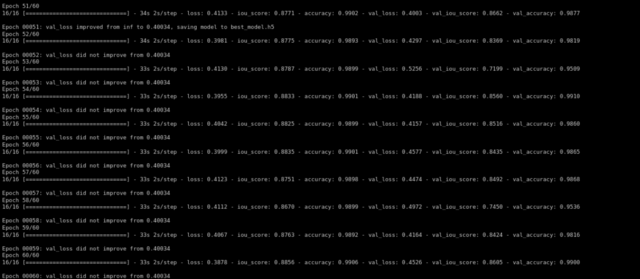
Nach dem Training für 60 Epochen, Wir haben eine Bestätigung iou_score von 0.86.
Schauen wir uns an, wie unser Modell funktionierte. Unser Modell wird einfach die Maske vorhersagen. Um das Bild ohne Schädel zu erhalten, wir müssen es über das Raw-Bild legen, um das Bild ohne Schädel zu erhalten …
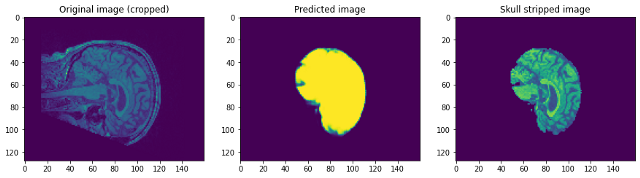
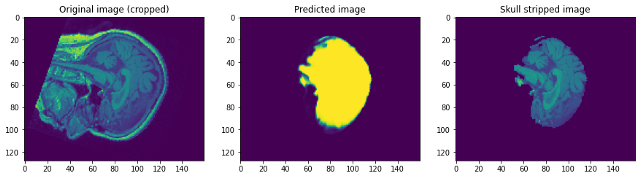

Wenn wir uns die Vorhersagen ansehen, können wir sagen, dass es zwar in der Lage ist, das Gehirn zu identifizieren und zu segmentieren, nicht annähernd perfekt. In diesem Punkt, Wir können uns mit einem Domänenexperten zusammensetzen, um herauszufinden, welche zusätzlichen Vorverarbeitungsschritte durchgeführt werden können, um die Genauigkeit zu verbessern. Aber was diesen Beitrag angeht, Ich schließe es hier ab. Bitte, Folgen link1 Ja / Ö link2 Wenn du mehr wissen willst …
Fazit:
Ich freue mich, dass du es bis zum Ende geschafft hast. Ich hoffe, dies hilft Ihnen beim Einstieg in die Bildsegmentierung in 3D-Bilder. Sie finden den Google Colab-Link, der den Code enthält. hier. Fühlen Sie sich frei, Vorschläge oder Fragen im Kommentarbereich hinzuzufügen. Einen schönen Tag noch!
Die in diesem Artikel zur Schädelentfernung gezeigten Medien sind nicht Eigentum von DataPeaker und werden nach Ermessen des Autors verwendet.






