Este artículo fue publicado como parte del Blogatón de ciencia de datos
La extracción del cráneo es uno de los pasos preliminares en el camino para detectar anomalías en el cerebro. Es el proceso de aislar tejido cerebral de tejido no cerebral a partir de una imagen de resonancia magnética de un cerebro. Esta segmentaciónLa segmentación es una técnica clave en marketing que consiste en dividir un mercado amplio en grupos más pequeños y homogéneos. Esta práctica permite a las empresas adaptar sus estrategias y mensajes a las características específicas de cada segmento, mejorando así la eficacia de sus campañas. La segmentación puede basarse en criterios demográficos, psicográficos, geográficos o conductuales, facilitando una comunicación más relevante y personalizada con el público objetivo.... del cerebro del cráneo es una tarea tediosa incluso para radiólogos expertos y la precisión de los resultados varía mucho de una persona a otra. Aquí estamos tratando de automatizar el proceso creando una canalización de extremo a extremo donde solo necesitamos ingresar la imagen de resonancia magnética sin procesar y la canalización debe generar una imagen segmentada del cerebro después de realizar el preprocesamiento necesario.
Entonces, ¿qué es una imagen de resonancia magnética?
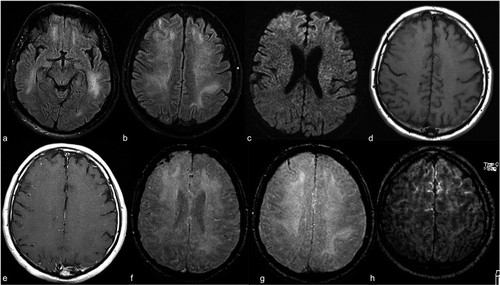
Para obtener una imagen de resonancia magnética de un paciente, se insertan en un túnel con un campo magnético en su interior. Esto hace que todos los protones del cuerpo se «alineen», por lo que su giro cuántico es el mismo. A continuación, se utiliza un pulso del campo magnético oscilante para interrumpir esta alineación. Cuando los protones vuelven al equilibrio, envían una onda electromagnética. En función del contenido de grasa, la composición química y, lo que es más importante, el tipo de estimulación (es decir, las secuencias) utilizadas para interrumpir los protones, se obtendrán diferentes imágenes. Cuatro secuencias comunes que se obtienen son T1, T1 con contraste (T1C), T2, y INSTINTO.
Desafíos comunes al trabajar con imágenes cerebrales
-
Desafíos de los datos del mundo real
Construir un modelo y lograr una buena precisión en una computadora portátil jupyter es bueno. Pero la mayoría de las veces, un modelo de muy buen rendimiento se comporta muy mal en datos del mundo real. Esto sucede debido a la deriva de datos cuando el modelo ve datos completamente diferentes a los que está entrenado. En nuestro caso, puede suceder debido a diferencias en algunos parámetrosLos "parámetros" son variables o criterios que se utilizan para definir, medir o evaluar un fenómeno o sistema. En diversos campos como la estadística, la informática y la investigación científica, los parámetros son fundamentales para establecer normas y estándares que guían el análisis y la interpretación de datos. Su adecuada selección y manejo son cruciales para obtener resultados precisos y relevantes en cualquier estudio o proyecto.... o métodos de generación de imágenes de resonancia magnética. Aquí hay un Blog que describe algunos fallos de la IA en el mundo real.
Formulación del problema
La tarea que tenemos aquí es dar una imagen de resonancia magnética en 3D que tenemos para identificar el cerebro y segmentar el tejido cerebral de la imagen completa de un cráneo. Para esta tarea, tendremos una etiqueta de verdad básica y, por lo tanto, será una tarea de segmentación de imágenes supervisada. Nosotros usaremos pérdida de dados como nuestra función de pérdidaLa función de pérdida es una herramienta fundamental en el aprendizaje automático que cuantifica la discrepancia entre las predicciones del modelo y los valores reales. Su objetivo es guiar el proceso de entrenamiento al minimizar esta diferencia, permitiendo así que el modelo aprenda de manera más efectiva. Existen diferentes tipos de funciones de pérdida, como el error cuadrático medio y la entropía cruzada, cada una adecuada para distintas tareas y....
Datos
Echemos un vistazo al conjunto de datos que usaremos para esta tarea. El conjunto de datos se puede descargar desde aquí.
El repositorio contiene datos de 125 participantes, de 21 a 45 años, con una variedad de síntomas psiquiátricos clínicos y subclínicos. Para cada participante, el repositorio contiene:
- Imagen de resonancia magnética ponderada en T1 estructural (sin rostro): esta es la imagen de resonancia magnética ponderada en T1 sin procesar con un solo canal.
- Máscara cerebral: es la máscara de imagen del cerebro o se puede llamar la verdad fundamental. Se obtiene utilizando el método Beast (extracción de cerebro basada en segmentación no local) y aplicando ediciones manuales por expertos en el dominio para eliminar tejido no cerebral.
- Imagen de cráneo despojado: esto se puede considerar como parte del cerebro despojado de la imagen ponderada en T1 anterior. Esto es similar a superponer máscaras a imágenes reales.
La resoluciónLa "resolución" se refiere a la capacidad de tomar decisiones firmes y cumplir con los objetivos establecidos. En contextos personales y profesionales, implica definir metas claras y desarrollar un plan de acción para alcanzarlas. La resolución es fundamental para el crecimiento personal y el éxito en diversas áreas de la vida, ya que permite superar obstáculos y mantener el enfoque en lo que realmente importa.... de las imágenes es de 1 mm3 y cada archivo está en formato NiFTI (.nii.gz). Un único punto de datos se parece a esto …
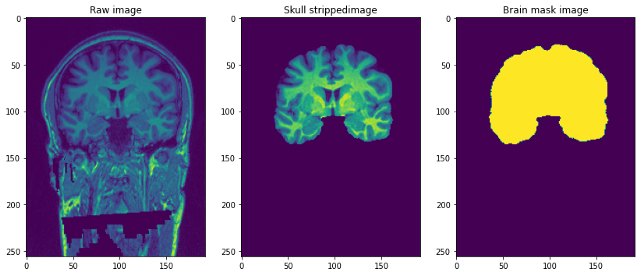
Preprocesando nuestras imágenes Raw
img=nib.load('/content/NFBS_Dataset/A00028185/sub-A00028185_ses-NFB3_T1w.nii.gz')
print('Shape of image=",img.shape)

Imagine imágenes de arriba en 3-D como si tuviéramos 192 imágenes en 2-D de tamaño 256 * 256 apiladas una encima de la otra.
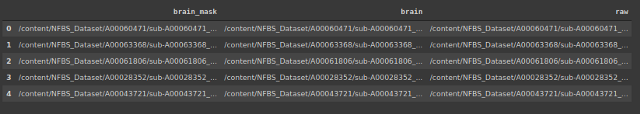
Creemos un marco de datos que contenga la ubicación de las imágenes y las máscaras correspondientes y las imágenes sin calaveras.
#storing the address of 3 types of files
import os
brain_mask=[]
brain=[]
raw=[]
for subdir, dirs, files in os.walk("/content/NFBS_Dataset'):
for file in files:
#print os.path.join(subdir, file)y
filepath = subdir + os.sep + file
if filepath.endswith(".gz"):
if '_brainmask.' in filepath:
brain_mask.append(filepath)
elif '_brain.' in filepath:
brain.append(filepath)
else:
raw.append(filepath)
La señal de campo de polarización es una señal de baja frecuencia y muy suave que corrompe las imágenes de resonancia magnética, especialmente las producidas por resonancias magnéticas antiguas (Imagen de resonancia magnética) máquinas. Los algoritmos de procesamiento de imágenes como la segmentación, el análisis de texturas o la clasificación que utilizan los valores de nivel de gris de los píxeles de la imagen no producirán resultados satisfactorios. Se necesita un paso de preprocesamiento para corregir la señal del campo de polarización antes de enviar imágenes de resonancia magnética corruptas a dichos algoritmos o los algoritmos deben modificarse.
-
Recortar y cambiar el tamaño
Debido a las limitaciones computacionales de ajustar la imagen completa al modelo aquí, decidimos reducir el tamaño de la imagen de resonancia magnética de (256 * 256 * 192) a (96 * 128 * 160). El tamaño del objetivo se elige de tal manera que se captura la mayor parte del cráneo y, después de recortarlo y redimensionarlo, tiene un efecto de centrado en las imágenes.
-
NormalizaciónLa normalización es un proceso fundamental en diversas disciplinas, que busca establecer estándares y criterios uniformes para mejorar la calidad y la eficiencia. En contextos como la ingeniería, la educación y la administración, la normalización facilita la comparación, la interoperabilidad y la comprensión mutua. Al implementar normas, se promueve la cohesión y se optimizan recursos, lo que contribuye al desarrollo sostenible y a la mejora continua de los procesos.... de la intensidad
La normalización cambia y escala una imagen para que los píxeles de la imagen tengan una media cero y una varianza unitaria. Esto ayuda a que el modelo converja más rápido al eliminar la variación de escala. A continuación se muestra el código para ello.
class preprocessing(): def __init__(self,df): self.data=df self.raw_index=[] self.mask_index=[] def bias_correction(self): !mkdir bias_correction n4 = N4BiasFieldCorrection() n4.inputs.dimension = 3 n4.inputs.shrink_factor = 3 n4.inputs.n_iterations = [20, 10, 10, 5] index_corr=[] for i in tqdm(range(len(self.data))): n4.inputs.input_image = self.data.raw.iloc[i] n4.inputs.output_image="bias_correction/"+str(i)+'.nii.gz' index_corr.append('bias_correction/'+str(i)+'.nii.gz') res = n4.run() index_corr=['bias_correction/'+str(i)+'.nii.gz' for i in range(125)] data['bias_corr']=index_corr print('Bias corrected images stored at : bias_correction/') def resize_crop(self): #Reducing the size of image due to memory constraints !mkdir resized target_shape = np.array((96,128,160)) #reducing size of image from 256*256*192 to 96*128*160 new_resolution = [2,]*3 new_affine = np.zeros((4,4)) new_affine[:3,:3] = np.diag(new_resolution) # putting point 0,0,0 in the middle of the new volume - this could be refined in the future new_affine[:3,3] = target_shape*new_resolution/2.*-1 new_affine[3,3] = 1. raw_index=[] mask_index=[] #resizing both image and mask and storing in folder for i in range(len(data)): downsampled_and_cropped_nii = resample_img(self.data.bias_corr.iloc[i], target_affine=new_affine, target_shape=target_shape, interpolation='nearest') downsampled_and_cropped_nii.to_filename('resized/raw'+str(i)+'.nii.gz') self.raw_index.append('resized/raw'+str(i)+'.nii.gz') downsampled_and_cropped_nii = resample_img(self.data.brain_mask.iloc[i], target_affine=new_affine, target_shape=target_shape, interpolation='nearest') downsampled_and_cropped_nii.to_filename('resized/mask'+str(i)+'.nii.gz') self.mask_index.append('resized/mask'+str(i)+'.nii.gz') return self.raw_index,self.mask_index def intensity_normalization(self): for i in self.raw_index: image = sitk.ReadImage(i) resacleFilter = sitk.RescaleIntensityImageFilter() resacleFilter.SetOutputMaximum(255) resacleFilter.SetOutputMinimum(0) image = resacleFilter.Execute(image) sitk.WriteImage(image,i) print('Normalization done. Images stored at: resized/')
Modelado
Ahora que nuestro preprocesamiento está terminado, podemos comenzar a modelar. Primero, haremos una prueba de tren dividida. Luego, usaremos un generador de datos personalizado para alimentar las imágenes de entrada al modelo.
Echemos un vistazo a la arquitectura del modelo.
def data_gen(self,img_list, mask_list, batch_size):
'''Custom data generator to feed image to model'''
c = 0
n = [i for i in range(len(img_list))] #List of training images
random.shuffle(n)
while (True):
img = np.zeros((batch_size, 96, 128, 160,1)).astype('float') #adding extra dimensions as conv3d takes file of size 5
mask = np.zeros((batch_size, 96, 128, 160,1)).astype('float')
for i in range(c, c+batch_size):
train_img = nib.load(img_list[n[i]]).get_data()
train_img=np.expand_dims(train_img,-1)
train_mask = nib.load(mask_list[n[i]]).get_data()
train_mask=np.expand_dims(train_mask,-1)
img[i-c]=train_img
mask[i-c] = train_mask
c+=batch_size
if(c+batch_size>=len(img_list)):
c=0
random.shuffle(n)
yield img,mask
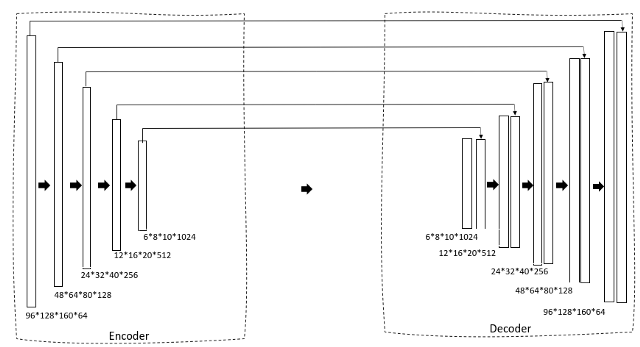
Estamos utilizando una U-Net 3D como nuestra arquitectura. Si ya está familiarizado con la U-Net 2D, esto será muy simple. Primero, tenemos una ruta de contracción a través de un codificador que reduce gradualmente el tamaño de la imagen y aumenta el número de filtros para generar características de cuello de botella. Esto luego se alimenta a un bloque de decodificador que expande gradualmente el tamaño para que finalmente pueda generar una máscara como la salida prevista.
def convolutional_block(input, filters=3, kernel_size=3, batchnorm = True):
'''conv layer followed by batchnormalization'''
x = Conv3D(filters = filters, kernel_size = (kernel_size, kernel_size,kernel_size),
kernel_initializer="he_normal", padding = 'same')(input)
if batchnorm:
x = BatchNormalization()(x)
x = Activation('relu')(x)
x = Conv3D(filters = filters, kernel_size = (kernel_size, kernel_size,kernel_size),
kernel_initializer="he_normal", padding = 'same')(input)
if batchnorm:
x = BatchNormalization()(x)
x = Activation('relu')(x)
return x
def resunet_opt(input_img, filters = 64, dropout = 0.2, batchnorm = True):
"""Residual 3D Unet"""
conv1 = convolutional_block(input_img, filters * 1, kernel_size = 3, batchnorm = batchnorm)
pool1 = MaxPooling3D((2, 2, 2))(conv1)
drop1 = Dropout(dropout)(pool1)
conv2 = convolutional_block(drop1, filters * 2, kernel_size = 3, batchnorm = batchnorm)
pool2 = MaxPooling3D((2, 2, 2))(conv2)
drop2 = Dropout(dropout)(pool2)
conv3 = convolutional_block(drop2, filters * 4, kernel_size = 3, batchnorm = batchnorm)
pool3 = MaxPooling3D((2, 2, 2))(conv3)
drop3 = Dropout(dropout)(pool3)
conv4 = convolutional_block(drop3, filters * 8, kernel_size = 3, batchnorm = batchnorm)
pool4 = MaxPooling3D((2, 2, 2))(conv4)
drop4 = Dropout(dropout)(pool4)
conv5 = convolutional_block(drop4, filters = filters * 16, kernel_size = 3, batchnorm = batchnorm)
conv5 = convolutional_block(conv5, filters = filters * 16, kernel_size = 3, batchnorm = batchnorm)
ups6 = Conv3DTranspose(filters * 8, (3, 3, 3), strides = (2, 2, 2), padding = 'same',activation='relu',kernel_initializer="he_normal")(conv5)
ups6 = concatenate([ups6, conv4])
ups6 = Dropout(dropout)(ups6)
conv6 = convolutional_block(ups6, filters * 8, kernel_size = 3, batchnorm = batchnorm)
ups7 = Conv3DTranspose(filters * 4, (3, 3, 3), strides = (2, 2, 2), padding = 'same',activation='relu',kernel_initializer="he_normal")(conv6)
ups7 = concatenate([ups7, conv3])
ups7 = Dropout(dropout)(ups7)
conv7 = convolutional_block(ups7, filters * 4, kernel_size = 3, batchnorm = batchnorm)
ups8 = Conv3DTranspose(filters * 2, (3, 3, 3), strides = (2, 2, 2), padding = 'same',activation='relu',kernel_initializer="he_normal")(conv7)
ups8 = concatenate([ups8, conv2])
ups8 = Dropout(dropout)(ups8)
conv8 = convolutional_block(ups8, filters * 2, kernel_size = 3, batchnorm = batchnorm)
ups9 = Conv3DTranspose(filters * 1, (3, 3, 3), strides = (2, 2, 2), padding = 'same',activation='relu',kernel_initializer="he_normal")(conv8)
ups9 = concatenate([ups9, conv1])
ups9 = Dropout(dropout)(ups9)
conv9 = convolutional_block(ups9, filters * 1, kernel_size = 3, batchnorm = batchnorm)
outputs = Conv3D(1, (1, 1, 2), activation='sigmoid',padding='same')(conv9)
model = Model(inputs=[input_img], outputs=[outputs])
return model
Luego entrenamos el modelo usando el optimizador de Adam y la pérdida de dados como nuestra función de pérdida …
def training(self,epochs):
im_height=96
im_width=128
img_depth=160
epochs=60
train_gen = data_gen(self.X_train,self.y_train, batch_size = 4)
val_gen = data_gen(self.X_test,self.y_test, batch_size = 4)
channels=1
input_img = Input((im_height, im_width,img_depth,channels), name="img")
self.model = resunet_opt(input_img, filters=16, dropout=0.05, batchnorm=True)
self.model.summary()
self.model.compile(optimizer=Adam(lr=1e-1),loss=focal_loss,metrics=[iou_score,'accuracy'])
#fitting the model
callbacks=callbacks = [
ModelCheckpoint('best_model.h5', verbose=1, save_best_only=True, save_weights_only=False)]
result=self.model.fit(train_gen,steps_per_epoch=16,epochs=epochs,validation_data=val_gen,validation_steps=16,initial_epoch=0,callbacks=callbacks)
Después de entrenar durante 60 épocas, obtuvimos un iou_score de validación de 0.86.
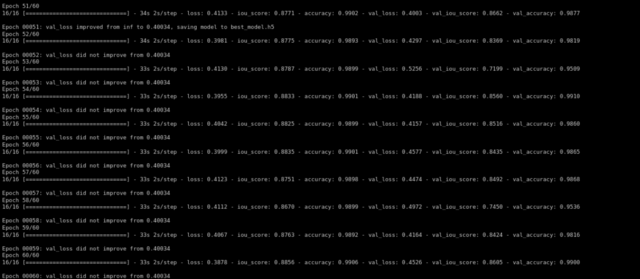
Echemos un vistazo a cómo funcionó nuestro modelo. Nuestro modelo predecirá simplemente la máscara. Para obtener la imagen sin calavera, necesitamos superponerla en la imagen Raw para obtener la imagen sin calavera …
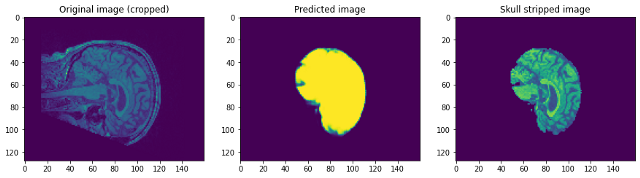
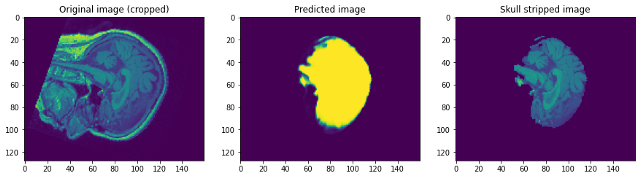

Mirando las predicciones podemos decir que aunque es capaz de identificar el cerebro y segmentarlo, no se acerca a la perfección. En este punto, podemos sentarnos con un experto en el dominio para identificar qué pasos adicionales de preprocesamiento se pueden realizar para mejorar la precisión. Pero en cuanto a este post, lo concluiré aquí. Por favor, siga enlace1 y / o enlace2 si quieres saber mas …
Conclusión:
Me alegro de que hayas llegado al final. Espero que esto te ayude a comenzar con la segmentación de imágenes en imágenes 3D. Puede encontrar el enlace de Google Colab que contiene el código. aquí. No dude en agregar sugerencias o consultas en la sección de comentarios. ¡Que tenga un lindo día!
Los medios que se muestran en este artículo sobre la eliminación del cráneo no son propiedad de DataPeaker y se utilizan a discreción del autor.






